
园艺学报 ›› 2025, Vol. 52 ›› Issue (3): 603-622.doi: 10.16420/j.issn.0513-353x.2023-0974
孙廷珍1, 疏琴1, 马玮1, 史玉滋1, 张蒙1, 向成钢2, 薄凯亮1, 段颖1,*( ), 王长林1,*(
), 王长林1,*( )
)
收稿日期:2024-10-10
修回日期:2025-01-09
出版日期:2025-03-25
发布日期:2025-03-25
通讯作者:
基金资助:
SUN Tingzhen1, SHU Qin1, MA Wei1, SHI Yuzi1, ZHANG Meng1, XIANG Chenggang2, BO Kailiang1, DUAN Ying1,*( ), WANG Changlin1,*(
), WANG Changlin1,*( )
)
Received:2024-10-10
Revised:2025-01-09
Published:2025-03-25
Online:2025-03-25
摘要:
赤霉素氧化酶家族GA2ox、GA3ox和GA20ox是植物激素赤霉素合成代谢途径后期关键酶,具有调控植株茎秆伸长等重要作用。以印度南瓜(Cucurbita maxima)参考基因组数据为基础,对其赤霉素氧化酶(GAox)家族基因进行鉴定,对其理化性质、基因结构、染色体定位、系统发育及其近缘物种基因组间的共线性关系等进行了系统分析,并在不同蔓长的印度南瓜种质中研究了其组织特异性表达模式。共鉴定到印度南瓜基因组中13个GA2ox,9个GA3ox和12个GA20ox基因。系统发育研究表明CmaGAox聚类为4个主要分支,上游启动子区分布有多个光响应、逆境胁迫、激素响应以及生长发育调控元件。CmaGAox在印度南瓜中存在9对串联复制基因和10对大片段复制基因,其中只有1对基因(CmaGA3ox5和CmaGA3ox6)受正向选择(Ka/Ks > 1)。共线性分析显示印度南瓜CmaGAox与美洲南瓜(Cucurbita pepo)和中国南瓜(Cucurbita moschata)分别存在47和48对共线性基因对。CmaGAox在果实中特异性表达较强,但CmaGA2ox1和CmaGA2ox4在主茎中表达量较高,并且在短蔓印度南瓜Cma0806中的表达量显著高于长蔓印度南瓜Cma0807,表明二者在主蔓生长调控中可能具有重要作用。
孙廷珍, 疏琴, 马玮, 史玉滋, 张蒙, 向成钢, 薄凯亮, 段颖, 王长林. 印度南瓜GAox家族基因鉴定及其在不同蔓长种质中的表达[J]. 园艺学报, 2025, 52(3): 603-622.
SUN Tingzhen, SHU Qin, MA Wei, SHI Yuzi, ZHANG Meng, XIANG Chenggang, BO Kailiang, DUAN Ying, WANG Changlin. Identification and Phylogenetic Analysis of Gibberellin Oxidase Gene Family and Its Expression Pattern in Main Vine Growth in Cucurbita maxima[J]. Acta Horticulturae Sinica, 2025, 52(3): 603-622.
| 引物名称 Primer name | 上游引物(5′-3′) Forward primer | 下游引物(5′-3′) Reverse primer |
|---|---|---|
| CmaGA2ox1 | GGCGATAAGGGCTGGATCGAATATCTC | ACCACTTCGCCGCTCAATTTCTTC |
| CmaGA2ox2 | CTACAGGCTTGGAGCAACGA | GGCAACTCCCGATTCCAGAA |
| CmaGA2ox3 | TGGCGTTCCCATGGAA TTCA | TATTCAACCCAGCCCACGTC |
| CmaGA2ox4 | AGCACGGAATTGGGGTTTCT | CACCGGTAAGTTCCACCCAA |
| CmaGA2ox5 | TGAAGAAGCTGAGCTGCGAA | AGCTTGGAGTTCCGGACATG |
| CmaGA2ox6 | GTTGTGGACACCTGTTTGCC | ATTCAATGGTGGTGCCCCAA |
| CmaGA2ox7 | TCCCGAGATTTTCCGGTGTG | ACAGCCTACTCAATGCGCTT |
| CmaGA2ox8 | ACAGCCGTTTGAGCGTAAGA | ATCATCGGAGCCGTTGGA TC |
| CmaGA2ox9 | ATGGCTGAGGGGTTAGGGAT | ACCCAAGCTTATTGGAGGGC |
| CmaGA2ox10 | AAAACAGAGAGCTGGCCCTC | ATGGAGAGGAACCCATCGGA |
| CmaGA2ox11 | TGGCTGGAACTTACCGATGG | GTGGGTCTGAGGGTGACATG |
| CmaGA2ox12 | TTTGGGGAGCATTCTGACCC | ACGTAGAATGCAGACGGGTC |
| CmaGA2ox13 | CCAGCCGACCCATTTGGATA | TCCACCGCACACCTGAAAAT |
| CmaGA20ox1 | GTGTTCGTCGACAACCAATGGAGAC | TCTGAGTGAACCGGAGGAGCATC |
| CmaGA20ox2 | CGACCCAACATCCCTCACCATTCT | ATCAGGGTACTGTCTTGGCCCTT |
| CmaGA20ox5 | GTAGACAACCAATGGCGTCTCATTGC | ATGTCAGCTCTGTAATGGTTGAGAGTG |
| CmaGA20ox10 | GGCTTTCAAGTGTGCGTCGATGATC | TCAAGCAACGTTCCCCATGTGTAATC |
| CmaGA20ox12 | CATCGCATGACAAAGTGGTGAGAGC | ATGGCAAGATCTCGGAAACTGGGTTC |
| CmaGA3ox1 | GATCCTCCGCAGAACATGCTTCTCA | TGCTGCTTTGGTGGAGGATTGTC |
| CmaGA3ox4 | GATTCAACGTCATAGGTTCTCCGGTTG | GTGTCTGGTGAAGCATGGTGAAGA |
| CmEF1a | ACGGTGATGCTGGTATGGTTA | CATTGTTGTTGGTTGGCTTATT |
表1 qRT-PCR引物序列
Table 1 Primers of qRT-PCR
| 引物名称 Primer name | 上游引物(5′-3′) Forward primer | 下游引物(5′-3′) Reverse primer |
|---|---|---|
| CmaGA2ox1 | GGCGATAAGGGCTGGATCGAATATCTC | ACCACTTCGCCGCTCAATTTCTTC |
| CmaGA2ox2 | CTACAGGCTTGGAGCAACGA | GGCAACTCCCGATTCCAGAA |
| CmaGA2ox3 | TGGCGTTCCCATGGAA TTCA | TATTCAACCCAGCCCACGTC |
| CmaGA2ox4 | AGCACGGAATTGGGGTTTCT | CACCGGTAAGTTCCACCCAA |
| CmaGA2ox5 | TGAAGAAGCTGAGCTGCGAA | AGCTTGGAGTTCCGGACATG |
| CmaGA2ox6 | GTTGTGGACACCTGTTTGCC | ATTCAATGGTGGTGCCCCAA |
| CmaGA2ox7 | TCCCGAGATTTTCCGGTGTG | ACAGCCTACTCAATGCGCTT |
| CmaGA2ox8 | ACAGCCGTTTGAGCGTAAGA | ATCATCGGAGCCGTTGGA TC |
| CmaGA2ox9 | ATGGCTGAGGGGTTAGGGAT | ACCCAAGCTTATTGGAGGGC |
| CmaGA2ox10 | AAAACAGAGAGCTGGCCCTC | ATGGAGAGGAACCCATCGGA |
| CmaGA2ox11 | TGGCTGGAACTTACCGATGG | GTGGGTCTGAGGGTGACATG |
| CmaGA2ox12 | TTTGGGGAGCATTCTGACCC | ACGTAGAATGCAGACGGGTC |
| CmaGA2ox13 | CCAGCCGACCCATTTGGATA | TCCACCGCACACCTGAAAAT |
| CmaGA20ox1 | GTGTTCGTCGACAACCAATGGAGAC | TCTGAGTGAACCGGAGGAGCATC |
| CmaGA20ox2 | CGACCCAACATCCCTCACCATTCT | ATCAGGGTACTGTCTTGGCCCTT |
| CmaGA20ox5 | GTAGACAACCAATGGCGTCTCATTGC | ATGTCAGCTCTGTAATGGTTGAGAGTG |
| CmaGA20ox10 | GGCTTTCAAGTGTGCGTCGATGATC | TCAAGCAACGTTCCCCATGTGTAATC |
| CmaGA20ox12 | CATCGCATGACAAAGTGGTGAGAGC | ATGGCAAGATCTCGGAAACTGGGTTC |
| CmaGA3ox1 | GATCCTCCGCAGAACATGCTTCTCA | TGCTGCTTTGGTGGAGGATTGTC |
| CmaGA3ox4 | GATTCAACGTCATAGGTTCTCCGGTTG | GTGTCTGGTGAAGCATGGTGAAGA |
| CmEF1a | ACGGTGATGCTGGTATGGTTA | CATTGTTGTTGGTTGGCTTATT |
| 基因名称 Gene name | 基因ID Gene ID | 位置 Location | 氨基酸长 Number of amino acids | 正负链 Strand | 等电点 pI | 分子量/kD Molecular weight | 疏水性 Hydro- phobicity | 亚细胞定位 Subcellular localization |
|---|---|---|---|---|---|---|---|---|
| CmaGA20ox1 | CmaCh02G010210 | 6058004 ~ 6060196 | 378 | - | 6.17 | 43.18 | -0.42 | Mb |
| CmaGA20ox2 | CmaCh03G011090 | 7574527 ~ 7576330 | 364 | - | 7.10 | 41.07 | -0.31 | Mb |
| CmaGA20ox3 | CmaCh07G008110 | 3735328 ~ 3737605 | 369 | - | 6.57 | 42.00 | -0.41 | Mb |
| CmaGA20ox4 | CmaCh15G006420 | 3035007 ~ 3036820 | 351 | + | 6.73 | 39.48 | -0.20 | Mb |
| CmaGA20ox5 | CmaCh15G013930 | 8764141 ~ 8766571 | 361 | - | 5.85 | 40.96 | -0.28 | Mb |
| CmaGA20ox6 | CmaCh16G001620 | 703878 ~ 705410 | 386 | - | 6.25 | 44.19 | -0.43 | Nu |
| CmaGA20ox7 | CmaCh17G000020 | 9087 ~ 10692 | 336 | + | 5.45 | 38.17 | -0.36 | Mb |
| CmaGA20ox8 | CmaCh18G012060 | 9561682 ~ 9563098 | 385 | + | 5.36 | 43.50 | -0.32 | Chl |
| CmaGA20ox9 | CmaCh18G012070 | 9565859 ~ 9567931 | 295 | + | 6.12 | 33.49 | -0.43 | Chl |
| CmaGA20ox10 | CmaCh18G012080 | 9570626 ~ 9572255 | 384 | + | 5.33 | 43.27 | -0.42 | Cy |
| CmaGA20ox11 | CmaCh18G012090 | 9574376 ~ 9575714 | 386 | + | 5.47 | 43.32 | -0.39 | Cy |
| CmaGA20ox12 | CmaCh18G012100 | 9579614 ~ 9583282 | 427 | + | 5.82 | 48.00 | -0.31 | Cy |
| CmaGA3ox1 | CmaCh05G006480 | 3289026 ~ 3290688 | 370 | + | 6.75 | 41.32 | -0.10 | Er |
| CmaGA3ox2 | CmaCh05G006490 | 3293066 ~ 3294672 | 367 | + | 6.03 | 41.37 | -0.13 | Cy |
| CmaGA3ox3 | CmaCh05G006500 | 3297430 ~ 3298596 | 346 | + | 6.67 | 38.94 | -0.20 | Mb |
| CmaGA3ox4 | CmaCh05G006510 | 3304268 ~ 3307142 | 383 | + | 6.13 | 43.31 | -0.19 | Cy |
| CmaGA3ox5 | CmaCh05G006520 | 3320084 ~ 3321250 | 344 | + | 6.50 | 38.77 | -0.21 | Cy |
| CmaGA3ox6 | CmaCh05G006530 | 3325577 ~ 3326736 | 348 | + | 6.22 | 39.53 | -0.22 | Cy |
| CmaGA3ox7 | CmaCh08G006320 | 3573502 ~ 3574908 | 377 | + | 6.62 | 41.73 | -0.21 | Mb |
| CmaGA3ox8 | CmaCh12G003100 | 1581698 ~ 1584654 | 348 | + | 5.72 | 39.17 | -0.23 | Cy |
| CmaGA3ox9 | CmaCh17G008310 | 6527065 ~ 6528927 | 342 | + | 7.27 | 38.55 | -0.26 | Mb |
| CmaGA2ox1 | CmaCh03G013990 | 8817543 ~ 8818816 | 331 | + | 6.27 | 36.85 | -0.31 | Cy |
| CmaGA2ox2 | CmaCh04G000330 | 191407 ~ 194668 | 374 | - | 6.70 | 41.93 | -0.30 | Cy |
| CmaGA2ox3 | CmaCh04G006490 | 3321297 ~ 3322998 | 340 | - | 8.31 | 37.75 | -0.20 | Chl |
| CmaGA2ox4 | CmaCh04G009310 | 4804076 ~ 4807014 | 360 | + | 5.70 | 41.35 | -0.31 | Cy |
| CmaGA2ox5 | CmaCh07G001050 | 581638 ~ 582804 | 332 | - | 5.76 | 36.82 | -0.21 | Mit |
| CmaGA2ox6 | CmaCh08G004850 | 2762847 ~ 2764115 | 331 | + | 8.75 | 36.59 | -0.22 | Mb |
| CmaGA2ox7 | CmaCh13G005360 | 5582531 ~ 5584247 | 336 | - | 5.96 | 37.73 | -0.16 | Cy |
| CmaGA2ox8 | CmaCh13G007180 | 6423868 ~ 6426404 | 341 | - | 5.96 | 38.51 | -0.28 | Mb |
| CmaGA2ox9 | CmaCh14G009480 | 4909702 ~ 4911273 | 335 | + | 5.89 | 37.09 | -0.10 | Chl |
| CmaGA2ox10 | CmaCh16G004530 | 2269857 ~ 2271606 | 327 | + | 7.02 | 36.21 | -0.23 | Chl |
| CmaGA2ox11 | CmaCh16G008420 | 4911602 ~ 4915537 | 349 | + | 6.07 | 40.10 | -0.44 | Cy |
| CmaGA2ox12 | CmaCh17G010160 | 7383663 ~ 7385904 | 329 | - | 8.70 | 36.45 | -0.26 | Mb |
| CmaGA2ox13 | CmaCh18G004710 | 2653034 ~ 2655073 | 336 | + | 6.14 | 37.85 | -0.20 | Cy |
表2 印度南瓜GA2ox、GA3ox和GA20ox蛋白理化性质
Table 2 Information of GA2ox、GA3ox and GA20ox gene families in Cucurbita maxima genome
| 基因名称 Gene name | 基因ID Gene ID | 位置 Location | 氨基酸长 Number of amino acids | 正负链 Strand | 等电点 pI | 分子量/kD Molecular weight | 疏水性 Hydro- phobicity | 亚细胞定位 Subcellular localization |
|---|---|---|---|---|---|---|---|---|
| CmaGA20ox1 | CmaCh02G010210 | 6058004 ~ 6060196 | 378 | - | 6.17 | 43.18 | -0.42 | Mb |
| CmaGA20ox2 | CmaCh03G011090 | 7574527 ~ 7576330 | 364 | - | 7.10 | 41.07 | -0.31 | Mb |
| CmaGA20ox3 | CmaCh07G008110 | 3735328 ~ 3737605 | 369 | - | 6.57 | 42.00 | -0.41 | Mb |
| CmaGA20ox4 | CmaCh15G006420 | 3035007 ~ 3036820 | 351 | + | 6.73 | 39.48 | -0.20 | Mb |
| CmaGA20ox5 | CmaCh15G013930 | 8764141 ~ 8766571 | 361 | - | 5.85 | 40.96 | -0.28 | Mb |
| CmaGA20ox6 | CmaCh16G001620 | 703878 ~ 705410 | 386 | - | 6.25 | 44.19 | -0.43 | Nu |
| CmaGA20ox7 | CmaCh17G000020 | 9087 ~ 10692 | 336 | + | 5.45 | 38.17 | -0.36 | Mb |
| CmaGA20ox8 | CmaCh18G012060 | 9561682 ~ 9563098 | 385 | + | 5.36 | 43.50 | -0.32 | Chl |
| CmaGA20ox9 | CmaCh18G012070 | 9565859 ~ 9567931 | 295 | + | 6.12 | 33.49 | -0.43 | Chl |
| CmaGA20ox10 | CmaCh18G012080 | 9570626 ~ 9572255 | 384 | + | 5.33 | 43.27 | -0.42 | Cy |
| CmaGA20ox11 | CmaCh18G012090 | 9574376 ~ 9575714 | 386 | + | 5.47 | 43.32 | -0.39 | Cy |
| CmaGA20ox12 | CmaCh18G012100 | 9579614 ~ 9583282 | 427 | + | 5.82 | 48.00 | -0.31 | Cy |
| CmaGA3ox1 | CmaCh05G006480 | 3289026 ~ 3290688 | 370 | + | 6.75 | 41.32 | -0.10 | Er |
| CmaGA3ox2 | CmaCh05G006490 | 3293066 ~ 3294672 | 367 | + | 6.03 | 41.37 | -0.13 | Cy |
| CmaGA3ox3 | CmaCh05G006500 | 3297430 ~ 3298596 | 346 | + | 6.67 | 38.94 | -0.20 | Mb |
| CmaGA3ox4 | CmaCh05G006510 | 3304268 ~ 3307142 | 383 | + | 6.13 | 43.31 | -0.19 | Cy |
| CmaGA3ox5 | CmaCh05G006520 | 3320084 ~ 3321250 | 344 | + | 6.50 | 38.77 | -0.21 | Cy |
| CmaGA3ox6 | CmaCh05G006530 | 3325577 ~ 3326736 | 348 | + | 6.22 | 39.53 | -0.22 | Cy |
| CmaGA3ox7 | CmaCh08G006320 | 3573502 ~ 3574908 | 377 | + | 6.62 | 41.73 | -0.21 | Mb |
| CmaGA3ox8 | CmaCh12G003100 | 1581698 ~ 1584654 | 348 | + | 5.72 | 39.17 | -0.23 | Cy |
| CmaGA3ox9 | CmaCh17G008310 | 6527065 ~ 6528927 | 342 | + | 7.27 | 38.55 | -0.26 | Mb |
| CmaGA2ox1 | CmaCh03G013990 | 8817543 ~ 8818816 | 331 | + | 6.27 | 36.85 | -0.31 | Cy |
| CmaGA2ox2 | CmaCh04G000330 | 191407 ~ 194668 | 374 | - | 6.70 | 41.93 | -0.30 | Cy |
| CmaGA2ox3 | CmaCh04G006490 | 3321297 ~ 3322998 | 340 | - | 8.31 | 37.75 | -0.20 | Chl |
| CmaGA2ox4 | CmaCh04G009310 | 4804076 ~ 4807014 | 360 | + | 5.70 | 41.35 | -0.31 | Cy |
| CmaGA2ox5 | CmaCh07G001050 | 581638 ~ 582804 | 332 | - | 5.76 | 36.82 | -0.21 | Mit |
| CmaGA2ox6 | CmaCh08G004850 | 2762847 ~ 2764115 | 331 | + | 8.75 | 36.59 | -0.22 | Mb |
| CmaGA2ox7 | CmaCh13G005360 | 5582531 ~ 5584247 | 336 | - | 5.96 | 37.73 | -0.16 | Cy |
| CmaGA2ox8 | CmaCh13G007180 | 6423868 ~ 6426404 | 341 | - | 5.96 | 38.51 | -0.28 | Mb |
| CmaGA2ox9 | CmaCh14G009480 | 4909702 ~ 4911273 | 335 | + | 5.89 | 37.09 | -0.10 | Chl |
| CmaGA2ox10 | CmaCh16G004530 | 2269857 ~ 2271606 | 327 | + | 7.02 | 36.21 | -0.23 | Chl |
| CmaGA2ox11 | CmaCh16G008420 | 4911602 ~ 4915537 | 349 | + | 6.07 | 40.10 | -0.44 | Cy |
| CmaGA2ox12 | CmaCh17G010160 | 7383663 ~ 7385904 | 329 | - | 8.70 | 36.45 | -0.26 | Mb |
| CmaGA2ox13 | CmaCh18G004710 | 2653034 ~ 2655073 | 336 | + | 6.14 | 37.85 | -0.20 | Cy |
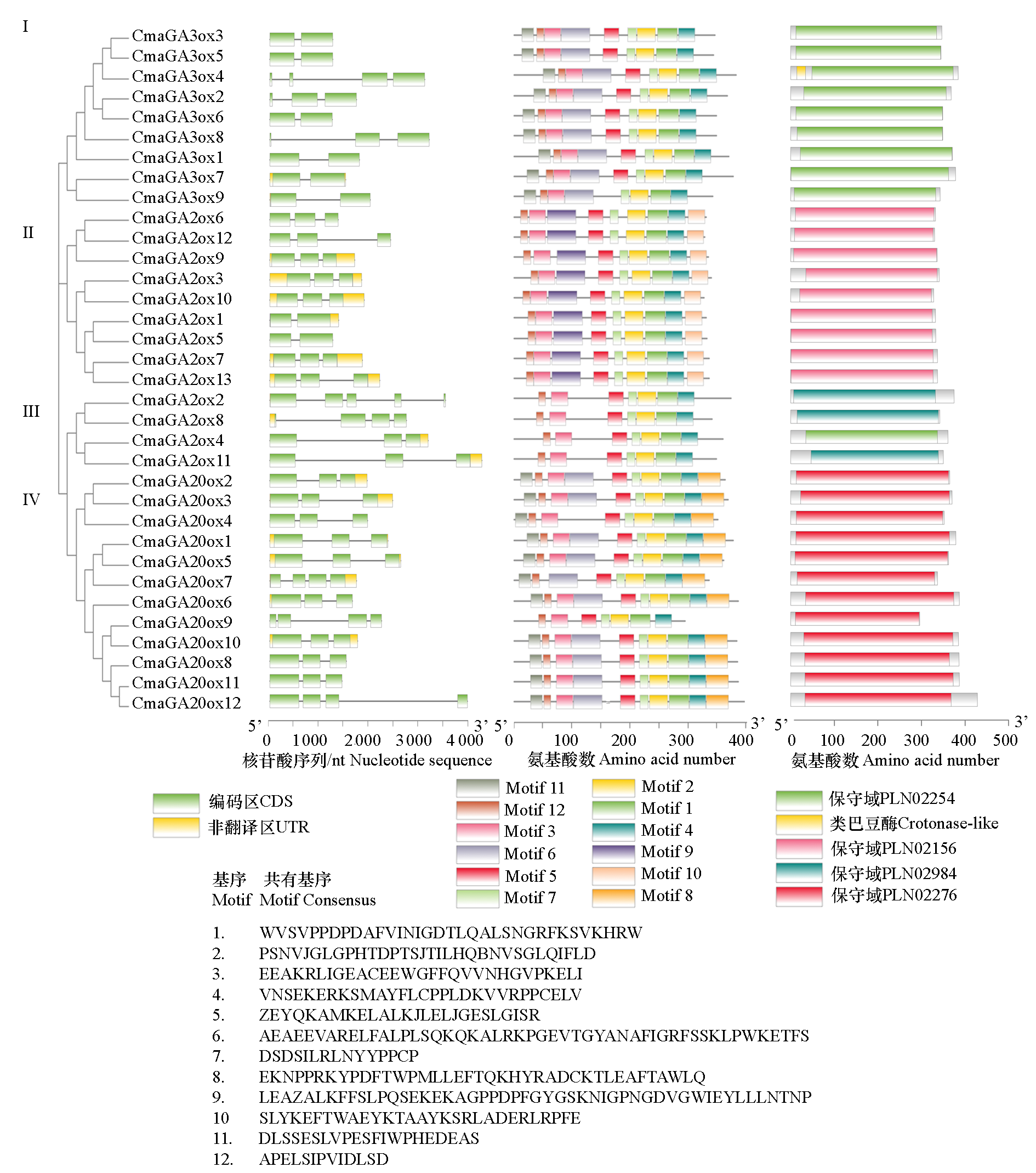
图3 印度南瓜GA2ox、GA3ox和GA20ox的基因结构、保守基序motif和保守结构域
Fig. 3 Gene structure,conserved motif and conserved domain of GA2ox,GA3ox and GA20ox in Cucurbita maxima I:GA3ox;II:C19 GA2ox;III:C20 GA2ox;IV:GA20ox

图4 不同植物GA2ox、GA3ox和GA20ox的系统进化树 At:拟南芥;Cma:印度南瓜;Pt:毛果杨;Cs:茶树;Md:苹果;Vv:葡萄;Citrus sinensis:甜橙;Gm:大豆;Mt:苜蓿;Cucumis sativus:黄瓜;Cla:西瓜;Sb:高粱;Zm:玉米;Os:水稻;Pe:毛竹
Fig. 4 Phylogenetic tree of GA2ox,GA3ox and GA20ox in different plants At:Arabidopsis;Cma:Cucurbita maxima;Pt:Populus trichocarpa;Cs:Camellia sinensis;Md:Malus × domestica;Vv:Vitis vinifera;Citrus sinensis:Sweet orange;Gm:Glycine max;Mt:Medicago truncatula;Cucumis sativus:Cucumber;Cla:Citrullus lanatus;Sb:Sorghum bicolor;Zm:Zea mays;Os:Oryza sativa;Pe:Phyllostachys edulis
| 串联复制基因对 Tandem duplication | Ka | Ks | Ka/Ks | 纯化选择 Purifying selection | 分化时间/Mya Divergence time |
|---|---|---|---|---|---|
| CmaGA20ox8/CmaGA20ox9 | 0.092 | 0.231 | 0.402 | 是 Yes | 7.70 |
| CmaGA20ox9/CmaGA20ox10 | 0.101 | 0.171 | 0.594 | 是 Yes | 5.69 |
| CmaGA20ox10/CmaGA20ox11 | 0.177 | 0.210 | 0.846 | 是 Yes | 6.99 |
| CmaGA20ox11/CmaGA20ox12 | 0.024 | 0.056 | 0.424 | 是 Yes | 1.87 |
| CmaGA3ox1/CmaGA3ox2 | 0.516 | 1.760 | 0.293 | 是 Yes | 58.65 |
| CmaGA3ox2/CmaGA3ox3 | 0.120 | 0.165 | 0.727 | 是 Yes | 5.50 |
| CmaGA3ox3/CmaGA3ox4 | 0.051 | 0.073 | 0.700 | 是 Yes | 2.45 |
| CmaGA3ox4/CmaGA3ox5 | 0.054 | 0.079 | 0.693 | 是 Yes | 2.62 |
| CmaGA3ox5/CmaGA3ox6 | 0.117 | 0.107 | 1.086 | 否 No | 3.58 |
表3 印度南瓜GA2ox、GA3ox、GA20ox家族基因发生串联复制的Ka/Ks比率及预测分化时间
Table 3 The Ka/Ks ratios and estimated divergence time for tandem duplication in Cucurbita maxima
| 串联复制基因对 Tandem duplication | Ka | Ks | Ka/Ks | 纯化选择 Purifying selection | 分化时间/Mya Divergence time |
|---|---|---|---|---|---|
| CmaGA20ox8/CmaGA20ox9 | 0.092 | 0.231 | 0.402 | 是 Yes | 7.70 |
| CmaGA20ox9/CmaGA20ox10 | 0.101 | 0.171 | 0.594 | 是 Yes | 5.69 |
| CmaGA20ox10/CmaGA20ox11 | 0.177 | 0.210 | 0.846 | 是 Yes | 6.99 |
| CmaGA20ox11/CmaGA20ox12 | 0.024 | 0.056 | 0.424 | 是 Yes | 1.87 |
| CmaGA3ox1/CmaGA3ox2 | 0.516 | 1.760 | 0.293 | 是 Yes | 58.65 |
| CmaGA3ox2/CmaGA3ox3 | 0.120 | 0.165 | 0.727 | 是 Yes | 5.50 |
| CmaGA3ox3/CmaGA3ox4 | 0.051 | 0.073 | 0.700 | 是 Yes | 2.45 |
| CmaGA3ox4/CmaGA3ox5 | 0.054 | 0.079 | 0.693 | 是 Yes | 2.62 |
| CmaGA3ox5/CmaGA3ox6 | 0.117 | 0.107 | 1.086 | 否 No | 3.58 |
| 大片段复制基因对 Segmental duplication | Ka | Ks | Ka/Ks | 纯化选择 Purifying selection | 分化时间/Mya Divergence ime |
|---|---|---|---|---|---|
| CmaGA20ox1/CmaGA20ox5 | 0.085 | 0.553 | 0.154 | 是 Yes | 18.45 |
| CmaGA20ox2/CmaGA20ox3 | 0.082 | 0.872 | 0.094 | 是 Yes | 29.08 |
| CmaGA20ox6/CmaGA20ox8 | 0.185 | 0.462 | 0.400 | 是 Yes | 15.40 |
| CmaGA2ox1/CmaGA2ox13 | 0.262 | 1.876 | 0.140 | 是 Yes | 62.54 |
| CmaGA2ox1/CmaGA2ox6 | 0.515 | NaN | NaN | 否 No | — |
| CmaGA2ox6/CmaGA2ox13 | 0.448 | 2.994 | 0.149 | 是 Yes | 99.80 |
| CmaGA2ox3/CmaGA2ox10 | 0.047 | 0.533 | 0.089 | 是 Yes | 17.77 |
| CmaGA2ox4/CmaGA2ox11 | 0.116 | 0.416 | 0.280 | 是 Yes | 13.87 |
| CmaGA2ox12/CmaGA2ox5 | 0.538 | NaN | NaN | 否 No | — |
| CmaGA3ox1/CmaGA3ox8 | 0.429 | 1.563 | 0.274 | 是 Yes | 52.10 |
表4 印度南瓜GA2ox、GA3ox、GA20ox家族基因发生大片段基因复制的Ka/Ks比率及预测分化时间
Table 4 The Ka/Ks ratios and estimated divergence time for segmental duplication in Cucurbita maxima
| 大片段复制基因对 Segmental duplication | Ka | Ks | Ka/Ks | 纯化选择 Purifying selection | 分化时间/Mya Divergence ime |
|---|---|---|---|---|---|
| CmaGA20ox1/CmaGA20ox5 | 0.085 | 0.553 | 0.154 | 是 Yes | 18.45 |
| CmaGA20ox2/CmaGA20ox3 | 0.082 | 0.872 | 0.094 | 是 Yes | 29.08 |
| CmaGA20ox6/CmaGA20ox8 | 0.185 | 0.462 | 0.400 | 是 Yes | 15.40 |
| CmaGA2ox1/CmaGA2ox13 | 0.262 | 1.876 | 0.140 | 是 Yes | 62.54 |
| CmaGA2ox1/CmaGA2ox6 | 0.515 | NaN | NaN | 否 No | — |
| CmaGA2ox6/CmaGA2ox13 | 0.448 | 2.994 | 0.149 | 是 Yes | 99.80 |
| CmaGA2ox3/CmaGA2ox10 | 0.047 | 0.533 | 0.089 | 是 Yes | 17.77 |
| CmaGA2ox4/CmaGA2ox11 | 0.116 | 0.416 | 0.280 | 是 Yes | 13.87 |
| CmaGA2ox12/CmaGA2ox5 | 0.538 | NaN | NaN | 否 No | — |
| CmaGA3ox1/CmaGA3ox8 | 0.429 | 1.563 | 0.274 | 是 Yes | 52.10 |

图8 印度南瓜授粉46 d后GA2ox、GA3ox和GA20ox的基因表达量 原始数据以log2(TPM+1)进行数据处理
Fig. 8 The gene expression of GA2ox、GA3ox and GA20ox at 46 days after pollination in Cucurbita maxima The original data are processed by log2(TPM+1)
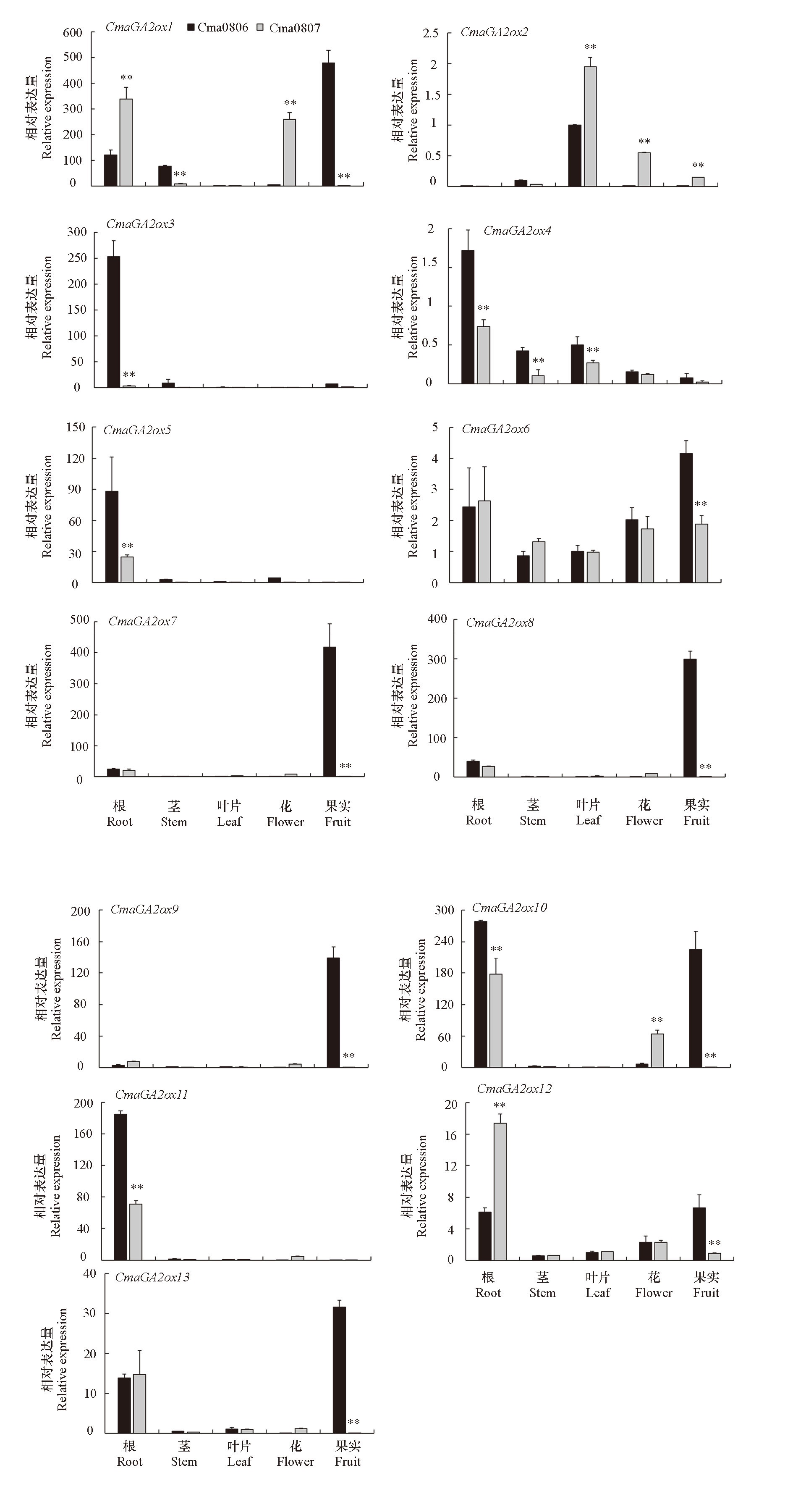
图9 印度南瓜GA2ox在短蔓种质Cma0806和长蔓种质Cma0807中的组织特异性表达 采用t检验对不同种质在同一组织中的基因表达数据进行显著性分析
Fig. 9 Tissue-specific expression analysis of GA2ox gene family members in dwarf-vine germplasm Cma0806 and long-vine germplasm Cma0807 in Cucurbita maxima Student’s t-test was used to perform the significance analysis of gene expression data of different germplasm in the same tissue

图10 印度南瓜GA3ox和GA20ox在短蔓种质Cma0806和长蔓种质Cma0807中的组织特异性表达 采用t检验对不同种质在同一组织中的基因表达数据进行显著性分析
Fig. 10 Tissue-specific expression analysis of GA3ox and GA20ox gene family members in dwarf-vine germplasm Cma0806 and long-vine germplasm Cma0807 in Cucurbita maxima Student’s t test was used to perform the significance analysis of gene expression data of different germplasm in the same tissue
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
|
董凤, 樊胜, 马小龙, 孟媛, 左希亚, 刘小杰, 李珂, 刘桢, 韩明玉, 张东. 2018. 苹果赤霉素氧化酶基因GA2ox、GA3ox和GA20ox家族全基因组鉴定及表达分析. 园艺学报, 45 (4):613-626.
doi: 10.16420/j.issn.0513-353x.2017-0452 |
|
| [7] |
doi: 10.1038/s41598-018-21293-1 pmid: 29440685 |
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
doi: 10.1016/s1360-1385(00)01790-8 pmid: 11120474 |
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
pmid: 8078921 |
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
doi: 10.1006/meth.2001.1262 pmid: 11846609 |
| [23] |
|
|
刘港运, 段世享, 许娜娜, 郭姚淼, 豆峻岭, 杨森, 牛欢欢, 刘东明, 杨路明, 胡建斌, 朱华玉. 2024. 分子标记辅助构建甜瓜矮化基因Cmerecta近等基因系. 园艺学报, 51 (9):2048-2062.
|
|
| [24] |
|
| [25] |
doi: 10.1093/jxb/erv300 pmid: 26093022 |
| [26] |
|
| [27] |
doi: 10.1111/pbi.12860 pmid: 29112324 |
| [28] |
|
| [29] |
doi: 10.1038/nbt847 pmid: 12858182 |
| [30] |
doi: 10.7505/j.issn.1007-9084.2019.03.012 |
|
盛晨, 张艳欣, 于景印, 高媛, 黎冬华, 周瑢, 张秀荣, 王林海. 2019. 芝麻赤霉素合成相关基因鉴定与表达分析. 中国油料作物学报, 41 (3):399-408.
|
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
doi: 10.1073/pnas.96.8.4698 pmid: 10200325 |
| [37] |
|
| [38] |
doi: 10.1038/s41477-022-01297-6 pmid: 36509843 |
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
|
向成钢. 2019. 印度南瓜矮生基因CmDw-1图位克隆及美洲南瓜矮生基因定位[博士论文]. 北京: 中国农业大学.
|
|
| [43] |
|
| [44] |
|
| [45] |
|
|
杨意宏, 王思宁, 徐浩, 孙化雨, 赵韩生, 陈段芬, 高志民. 2018. 毛竹赤霉素合成相关酶基因的全基因组鉴定和表达分析. 基因组学与应用生物学, 37 (9):3966-3977.
|
|
| [46] |
|
| [47] |
|
| [48] |
doi: 10.11869/j.issn.100-8551.2019.06.1096 |
|
朱海生, 刘建汀, 温文旭, 李永平, 王彬, 陈敏氡, 温庆放. 2019. 印度南瓜延伸因子基因CmEF1a的克隆与分析. 核农学报,(6):1096-1104.
doi: 10.11869/j.issn.100-8551.2019.06.1096 |
| [1] | 邓淑琴, 高莹瑞, 李雨桐, 王瑛, 龚春梅, 白娟. 茶树泛素连接酶基因CsPUB21对非生物胁迫的响应[J]. 园艺学报, 2025, 52(3): 655-670. |
| [2] | 仝宗军, 韩星, 段昕莲, 刘媛媛, 林俊彬, 甘颖, 陈杰, 谢宝贵, 甘炳成, 严俊杰. 金针菇PRX家族基因鉴定及其在子实体发育过程中的表达分析[J]. 园艺学报, 2025, 52(2): 337-348. |
| [3] | 王珊珊, 郭瑞, 何棱, 吴春红, 陈禅友, 万何平, 赵慧霞. 长豇豆Lhc家族基因鉴定及其在盐胁迫下的表达分析[J]. 园艺学报, 2025, 52(1): 111-122. |
| [4] | 贺丹丹, 何宏泰, 王文庭, 周文美, 刘燕敏, 刘骕骦. 甜瓜GolS家族基因鉴定及其响应低温胁迫的表达分析[J]. 园艺学报, 2025, 52(1): 136-148. |
| [5] | 郭昌权, 李丹琪, 惠馨冉, 郑婧雅, 侯梦璐, 朱永兴. 姜DUF966基因家族成员鉴定与表达分析[J]. 园艺学报, 2024, 51(9): 2031-2047. |
| [6] | 赵佳莹, 曾周婷, 岑欣颖, 施姣淇, 李效贤, 沈晓霞, 俞振明. 铁皮石斛CCO基因家族鉴定及其在花发育中的表达分析[J]. 园艺学报, 2024, 51(9): 2075-2088. |
| [7] | 王艳红, 席克勇, 田野, 刘德麒, 周克贵, 尹军良, 刘奕清, 朱永兴. 姜GST基因家族成员鉴定与表达分析[J]. 园艺学报, 2024, 51(8): 1803-1822. |
| [8] | 陈佳悦, 段英明, 周雁, 肖扬, 边银丙, 龚钰华. 香菇ALDH家族基因鉴定、表达及功能分析[J]. 园艺学报, 2024, 51(5): 1033-1046. |
| [9] | 刘艳艳, 丁颖, 刘兴华, 郑佳秋, 刘志钦. 辣椒CaSYT1的鉴定及其在疫霉侵染过程中的功能初探[J]. 园艺学报, 2024, 51(3): 533-544. |
| [10] | 刘晋红, 王峥, 于昊, 辛依睿, 亓果宁, 柳参奎, 任慧敏. 毛竹SLAC家族基因鉴定及PheSLAC1功能分析[J]. 园艺学报, 2024, 51(3): 545-559. |
| [11] | 罗新锐, 张晓旭, 王玉萍, 王智, 马媛媛, 周丙月. 普通菜豆Trihelix基因家族鉴定及表达分析[J]. 园艺学报, 2024, 51(12): 2775-2790. |
| [12] | 李宇腾, 陈瑶, 任恒泽, 李聪聪, 王浩乾, 曹红利, 岳川, 郝心愿, 王新超. 茶树CsIDM的鉴定、表达分析及互作验证[J]. 园艺学报, 2023, 50(8): 1679-1696. |
| [13] | 薛珍珍, 关鸿发, 李娜, 李凌飞, 钟春梅. 铁十字秋海棠GASA家族全基因组鉴定及叶斑形成的初步探索[J]. 园艺学报, 2023, 50(7): 1482-1494. |
| [14] | 黄治皓, 刘婷婷, 董旭杰, 严明理, 刘志祥, 曾超珍. 芥菜HMA家族基因鉴定及其在镉胁迫下的表达分析[J]. 园艺学报, 2023, 50(6): 1230-1242. |
| [15] | 王同欢, 吴雨馨, 武艺圆, 李鑫鑫, 刘梦阳, 杨莲莲, 李佳鹏, 张忠山, 曹访, 仲雪婷, 王占旗. 菊花脑GRAS家族鉴定及其低温胁迫响应表达分析[J]. 园艺学报, 2023, 50(4): 815-830. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2012 《园艺学报》编辑部 京ICP备10030308号-2 国际联网备案号 11010802023439
编辑部地址: 北京市海淀区中关村南大街12号中国农业科学院蔬菜花卉研究所 邮编: 100081
电话: 010-82109523 E-Mail: yuanyixuebao@126.com
技术支持:北京玛格泰克科技发展有限公司