
园艺学报 ›› 2024, Vol. 51 ›› Issue (8): 1803-1822.doi: 10.16420/j.issn.0513-353x.2023-0384
王艳红1, 席克勇1, 田野1, 刘德麒1, 周克贵1, 尹军良2, 刘奕清1, 朱永兴1,*( )
)
收稿日期:2024-03-22
修回日期:2024-05-17
出版日期:2024-08-25
发布日期:2024-08-21
通讯作者:
基金资助:
WANG Yanhong1, XI Keyong1, TIAN Ye1, LIU Deqi1, ZHOU Kegui1, YIN Junliang2, LIU Yiqing1, ZHU Yongxing1,*( )
)
Received:2024-03-22
Revised:2024-05-17
Published:2024-08-25
Online:2024-08-21
摘要:
为解析姜(Zingiber officinale Roscoe)谷胱甘肽硫转移酶(glutathione-S-transferase,GST)基因家族的信息,基于姜基因组数据,分析鉴定了ZoGST家族成员及其表达模式,并进一步利用qRT-PCR检测了10个ZoGST在不同胁迫下的表达特性。在姜基因组中共鉴定到58个GST家族成员,其编码蛋白的氨基酸长度在110 ~ 675 aa之间,分子量12.21 ~ 72.51 kD,等电点4.55 ~ 9.51。系统进化树显示,ZoGST可分为Ⅰ~Ⅹ亚家族,第Ⅶ和Ⅹ亚家族成员居多数;ZoGST家族成员分布在11条染色体上,共线性分析表明该家族成员存在6对片段重复;顺式作用调控元件分析发现上游启动子区含有与光响应、逆境胁迫、激素调节相关的作用元件;转录组测序数据分析显示,ZoGST有一定的组织表达特异性且响应病害、低温等逆境胁迫,其中,ZoGSTDHAR5、ZoGSTF9、ZoGSTF13等在姜不同生长时期、不同部位、低温和病害胁迫下均有较高表达。qRT-PCR分析表明,盐胁迫下ZoGSTDHAR2(除根茎外)、ZoGSTEF1G2、ZoGSTF9在姜不同组织表达量均显著下调;淹水胁迫下,ZoGSTDHAR2等在叶和根茎中表达量显著上调;干旱胁迫下,ZoGSTF9等在根中表达量显著上调。
王艳红, 席克勇, 田野, 刘德麒, 周克贵, 尹军良, 刘奕清, 朱永兴. 姜GST基因家族成员鉴定与表达分析[J]. 园艺学报, 2024, 51(8): 1803-1822.
WANG Yanhong, XI Keyong, TIAN Ye, LIU Deqi, ZHOU Kegui, YIN Junliang, LIU Yiqing, ZHU Yongxing. Identification and Expression Pattern Analysis of GST in Zingiber officinale[J]. Acta Horticulturae Sinica, 2024, 51(8): 1803-1822.
| 登录号 Accession ID | 处理样品描述 Description | 数据大小 Data size | 参考文献 Reference |
|---|---|---|---|
| PRJNA788194 | 不同发育阶段根茎、不同组织 Different developmental stages of rhizomes,different tissues | 未知 Unknown | Li et al., |
| PRJNA592215 | 红茎内部组织、绿茎、黄根茎、红茎表皮 Internal tissue of red stem,green stem,yellow rhizome,red stem epidermis | 254 Mb | Li et al., |
| PRJNA380847 | 姜根茎低/高土壤湿度下用无菌水和青枯菌接种 Inoculate ginger rhizomes with sterile water and Ralstonia solanacearum under low/high soil moisture | 未知 Unknown | Jiang et al., |
| PRJNA380972 | 高土壤湿度增加姜中青枯菌感染率 High-soil-moisture-elevated susceptibility to R. solanacearum infection in ginger | 31.5 Gb | Huang et al., |
| PRJNA911443 | 姜采收后26、10和2 ℃贮藏条件下的根茎 Rhizome of ginger harvested under storage conditions of 26,10,and 2 ℃ | 16.8 Gb | Zhang et al., |
表1 姜各转录组数据集来源
Table 1 Source of data sets of transcriptome of ginger
| 登录号 Accession ID | 处理样品描述 Description | 数据大小 Data size | 参考文献 Reference |
|---|---|---|---|
| PRJNA788194 | 不同发育阶段根茎、不同组织 Different developmental stages of rhizomes,different tissues | 未知 Unknown | Li et al., |
| PRJNA592215 | 红茎内部组织、绿茎、黄根茎、红茎表皮 Internal tissue of red stem,green stem,yellow rhizome,red stem epidermis | 254 Mb | Li et al., |
| PRJNA380847 | 姜根茎低/高土壤湿度下用无菌水和青枯菌接种 Inoculate ginger rhizomes with sterile water and Ralstonia solanacearum under low/high soil moisture | 未知 Unknown | Jiang et al., |
| PRJNA380972 | 高土壤湿度增加姜中青枯菌感染率 High-soil-moisture-elevated susceptibility to R. solanacearum infection in ginger | 31.5 Gb | Huang et al., |
| PRJNA911443 | 姜采收后26、10和2 ℃贮藏条件下的根茎 Rhizome of ginger harvested under storage conditions of 26,10,and 2 ℃ | 16.8 Gb | Zhang et al., |
| 基因名 Gene name | 正向引物(5′-3′) Forward primer | 反向引物(5′-3′) Reverse primer | 长度/bp Length |
|---|---|---|---|
| ZoGSTDHAR2 | CTCCTCCTGCCATTTCCTCG | GATTCACCTCCACGACTTTGTATG | 228 |
| ZoGSTEF1G2 | AAGGTGTTAGGCGAGGTCAAG | TGGGTGCTTCTTCCTCTTCTTC | 169 |
| ZoGSTCHQ1 | GGTCACAGAATGGGTGGAGAG | TGGACCATCCGAGCAATCAC | 129 |
| ZoGSTT1 | AGAGTCATGCCATATTGAGCTACC | ATTGAACCACGGCGTAGATTG | 140 |
| ZoGSTL1 | CTTCCTCCGTCCCTCGATTC | GTTATCCAGGTGCGTTGTGC | 107 |
| ZoGSTL2 | TCCCGCTTTGTGAAGGAGG | CGCAAATGGGCAGGTGTATG | 111 |
| ZoGSTF9 | GGTGGTACGACGGACGAGAAG | TGACGGGATAGTGGCAGAGG | 145 |
| ZoGSTU13 | TTGCTGGATCTGTGGGTGAGTC | GTATTGCACGATGATGAGCGACT | 201 |
| ZoGSTU16 | TGGATCTGTGGGTGAGTCCTTTC | TGTTGTCCAAGTCCTGCTCCTG | 96 |
| ZoGSTU27 | GGTGTTACTGGATCTGTGGGTGAG | GCAACGGGCTCTTGTTGTCC | 116 |
| RBP | CCTATGAAGCGTAGAAACACAAG | GGAAGGACAACATCCCAAATC | 123 |
表2 ZoGST家族荧光定量PCR引物序列
Table 2 Primer sequences for fluorescence quantitative PCR of ZoGST
| 基因名 Gene name | 正向引物(5′-3′) Forward primer | 反向引物(5′-3′) Reverse primer | 长度/bp Length |
|---|---|---|---|
| ZoGSTDHAR2 | CTCCTCCTGCCATTTCCTCG | GATTCACCTCCACGACTTTGTATG | 228 |
| ZoGSTEF1G2 | AAGGTGTTAGGCGAGGTCAAG | TGGGTGCTTCTTCCTCTTCTTC | 169 |
| ZoGSTCHQ1 | GGTCACAGAATGGGTGGAGAG | TGGACCATCCGAGCAATCAC | 129 |
| ZoGSTT1 | AGAGTCATGCCATATTGAGCTACC | ATTGAACCACGGCGTAGATTG | 140 |
| ZoGSTL1 | CTTCCTCCGTCCCTCGATTC | GTTATCCAGGTGCGTTGTGC | 107 |
| ZoGSTL2 | TCCCGCTTTGTGAAGGAGG | CGCAAATGGGCAGGTGTATG | 111 |
| ZoGSTF9 | GGTGGTACGACGGACGAGAAG | TGACGGGATAGTGGCAGAGG | 145 |
| ZoGSTU13 | TTGCTGGATCTGTGGGTGAGTC | GTATTGCACGATGATGAGCGACT | 201 |
| ZoGSTU16 | TGGATCTGTGGGTGAGTCCTTTC | TGTTGTCCAAGTCCTGCTCCTG | 96 |
| ZoGSTU27 | GGTGTTACTGGATCTGTGGGTGAG | GCAACGGGCTCTTGTTGTCC | 116 |
| RBP | CCTATGAAGCGTAGAAACACAAG | GGAAGGACAACATCCCAAATC | 123 |
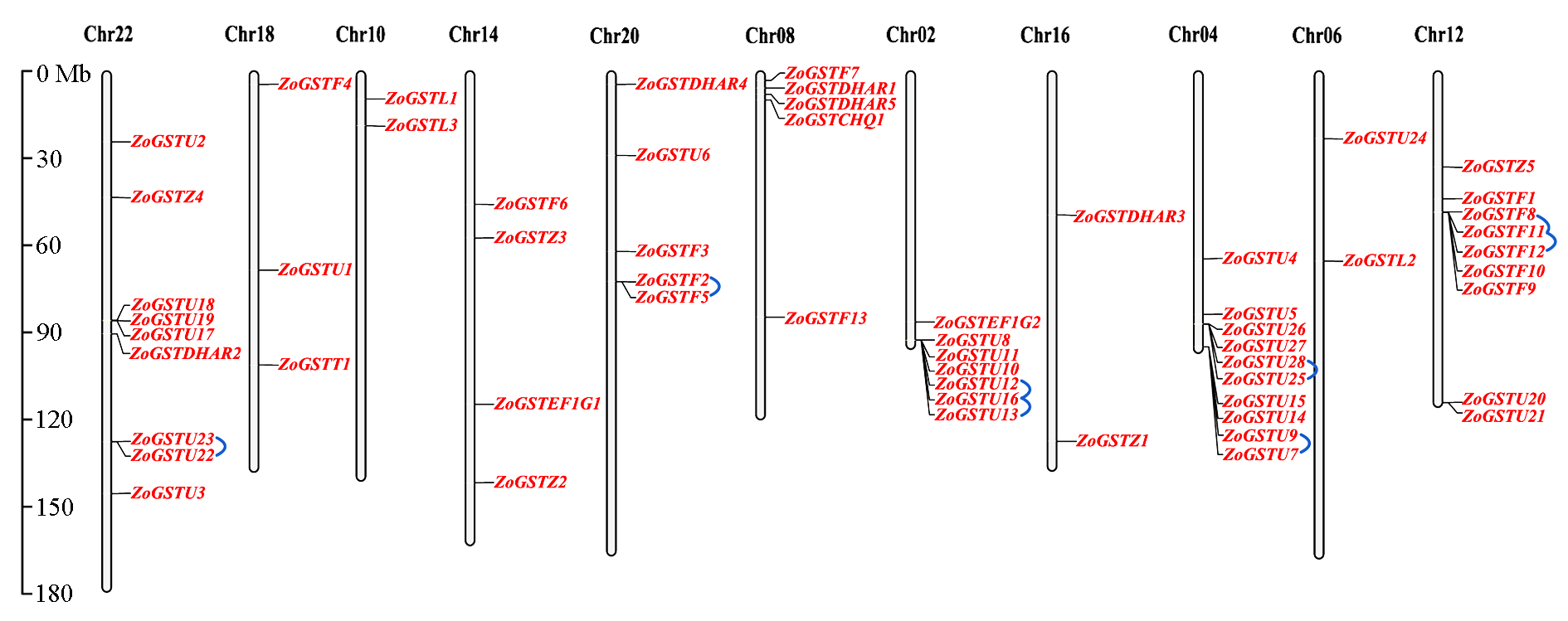
图2 ZoGST家族基因染色体分布 图中蓝色弧线代表基因间的串联重复。
Fig. 2 Chromosomal localization of ZoGST genes family The blue arc in the figure represents tandem repeats between genes.
| 基因1 | 基因2 | 异义替换频率(Ka) | 同义替换频率(Ks) | 异义替换/同义替换 |
|---|---|---|---|---|
| Gene 1 | Gene 2 | Non-synonymous | Synonymous | Ka/Ks |
| ZoGSTL1 | ZoGSTL3 | 0.141203891 | 0.527940985 | 0.267461505 |
| ZoGSTL2 | ZoGSTL3 | 0.096538384 | 0.315771474 | 0.305722310 |
| ZoGSTDHAR2 | ZoGSTDHAR3 | 0.059875159 | 0.365643255 | 0.163752943 |
| ZoGSTF2 | ZoGSTF4 | 0.389135669 | 0.971421814 | 0.400583622 |
| ZoGSTU2 | ZoGSTU3 | 0.113145323 | 0.259868329 | 0.435394813 |
| ZoGSTU11 | ZoGSTU14 | 0.073775974 | 0.133473189 | 0.552740031 |
表3 ZoGST基因的Ka/Ks
Table 3 Ka/Ks of ZoGST genes
| 基因1 | 基因2 | 异义替换频率(Ka) | 同义替换频率(Ks) | 异义替换/同义替换 |
|---|---|---|---|---|
| Gene 1 | Gene 2 | Non-synonymous | Synonymous | Ka/Ks |
| ZoGSTL1 | ZoGSTL3 | 0.141203891 | 0.527940985 | 0.267461505 |
| ZoGSTL2 | ZoGSTL3 | 0.096538384 | 0.315771474 | 0.305722310 |
| ZoGSTDHAR2 | ZoGSTDHAR3 | 0.059875159 | 0.365643255 | 0.163752943 |
| ZoGSTF2 | ZoGSTF4 | 0.389135669 | 0.971421814 | 0.400583622 |
| ZoGSTU2 | ZoGSTU3 | 0.113145323 | 0.259868329 | 0.435394813 |
| ZoGSTU11 | ZoGSTU14 | 0.073775974 | 0.133473189 | 0.552740031 |
| 名称 Name | 基因ID Gene ID | 长度/aa Length | 分子量/kD Mw | 等电点 pI | 不稳定指数 Instability index | 亲水性 Hydrophilicity | 亚细胞定位 Subcellular localization |
|---|---|---|---|---|---|---|---|
| ZoGSTDHAR1 | Maker00035113 | 330 | 37.20 | 5.06 | 51.08 | -0.278 | Cp,Cy,Mt |
| ZoGSTDHAR2 | Maker00052306 | 328 | 36.71 | 9.10 | 47.47 | -0.285 | Cp |
| ZoGSTDHAR3 | Maker00046027 | 350 | 39.34 | 8.79 | 44.65 | -0.139 | Mt |
| ZoGSTDHAR5 | Maker00035024 | 255 | 28.17 | 6.25 | 43.08 | -0.232 | Cp,Cy |
| ZoGSTDHAR4 | Maker00022334 | 267 | 29.40 | 6.91 | 48.36 | -0.098 | Cp,Cy |
| ZoGSTEF1G1 | Maker00033503 | 418 | 47.80 | 5.62 | 40.78 | -0.387 | Cy |
| ZoGSTEF1G2 | Maker00031875 | 419 | 47.96 | 5.68 | 39.85 | -0.346 | Cy |
| ZoGSTCHQ1 | Maker00046252 | 265 | 31.38 | 9.20 | 36.80 | -0.314 | Cy |
| ZoGSTT1 | Maker00029139 | 263 | 29.87 | 8.99 | 41.86 | -0.200 | Cy |
| ZoGSTL1 | Maker00033284 | 218 | 24.45 | 4.57 | 49.88 | -0.138 | Nu |
| ZoGSTL2 | Maker00059206 | 262 | 29.82 | 5.32 | 41.03 | -0.344 | Cp |
| ZoGSTL3 | Maker00009568 | 241 | 27.61 | 5.28 | 42.39 | -0.194 | Cy |
| ZoGSTZ1 | Maker00037272 | 346 | 39.86 | 6.47 | 34.33 | -0.469 | Mt |
| ZoGSTZ2 | Maker00013992 | 420 | 46.61 | 8.85 | 57.36 | -0.186 | Cy |
| ZoGSTZ3 | Maker00051190 | 490 | 55.36 | 4.71 | 53.11 | -0.735 | Cp |
| ZoGSTZ4 | Maker00021183 | 334 | 37.27 | 8.20 | 52.82 | -0.159 | Cp |
| ZoGSTZ5 | Maker00078047 | 402 | 44.92 | 5.87 | 44.01 | -0.161 | Cy |
| ZoGSTF1 | Maker00077732 | 675 | 72.51 | 5.62 | 60.97 | -0.818 | Nu |
| ZoGSTF2 | Maker00076791 | 220 | 24.34 | 6.91 | 55.02 | -0.311 | Cy |
| ZoGSTF3 | Maker00068242 | 318 | 35.47 | 6.56 | 54.65 | -0.470 | Cp,Cy,Nu |
| ZoGSTF4 | Maker00047494 | 221 | 25.42 | 7.76 | 50.63 | -0.422 | Cy |
| ZoGSTF5 | Maker00076803 | 221 | 25.31 | 6.97 | 55.37 | -0.513 | Cy |
| ZoGSTF6 | Maker00050934 | 243 | 27.74 | 6.34 | 39.92 | -0.216 | Cy |
| ZoGSTF7 | Maker00023234 | 224 | 25.10 | 5.35 | 41.26 | -0.144 | Cy |
| ZoGSTF8 | Maker00077970 | 251 | 28.38 | 6.72 | 38.38 | 0.051 | Cy |
| ZoGSTF9 | Maker00078922 | 217 | 24.39 | 5.52 | 49.34 | -0.089 | Cy |
| ZoGSTF10 | Maker00078286 | 217 | 24.47 | 5.71 | 37.72 | 0.043 | Cy |
| ZoGSTF11 | Maker00078094 | 255 | 28.60 | 6.31 | 39.33 | 0.025 | Cy |
| ZoGSTF12 | Maker00077516 | 219 | 24.50 | 5.53 | 36.69 | 0.050 | Cy |
| ZoGSTF13 | Maker00040344 | 217 | 23.76 | 5.97 | 18.75 | 0.107 | Cy |
| ZoGSTU1 | Maker00008855 | 222 | 24.81 | 5.39 | 39.79 | -0.027 | Cy |
| ZoGSTU2 | Maker00015777 | 244 | 27.20 | 5.02 | 38.10 | -0.195 | Cy |
| ZoGSTU3 | Maker00049005 | 247 | 27.51 | 5.40 | 34.94 | -0.146 | Cy |
| ZoGSTU4 | Maker00045495 | 232 | 26.80 | 6.20 | 55.74 | -0.338 | Cy |
| ZoGSTU5 | Maker00045023 | 228 | 26.25 | 5.47 | 48.11 | -0.321 | Cy |
| ZoGSTU6 | Maker00061695 | 227 | 25.65 | 5.49 | 55.78 | 0.027 | Cy |
| ZoGSTU7 | Maker00045310 | 222 | 24.36 | 5.43 | 24.27 | 0.064 | Cy |
| ZoGSTU8 | Maker00042846 | 222 | 24.37 | 5.43 | 23.19 | 0.060 | Cy |
| ZoGSTU9 | Maker00044620 | 219 | 24.93 | 5.29 | 34.73 | -0.096 | Cy |
| ZoGSTU10 | Maker00042772 | 219 | 24.91 | 5.39 | 34.62 | -0.086 | Cy |
| ZoGSTU11 | Maker00042767 | 117 | 13.53 | 5.26 | 31.22 | -0.222 | Cp |
| ZoGSTU12 | Maker00042721 | 252 | 28.66 | 5.40 | 37.67 | -0.124 | Cy |
| ZoGSTU13 | Maker00042760 | 216 | 24.66 | 5.15 | 34.00 | -0.111 | Cy |
| ZoGSTU14 | Maker00045562 | 252 | 28.79 | 5.28 | 38.35 | -0.142 | Cy |
| ZoGSTU15 | Maker00045539 | 216 | 24.59 | 5.25 | 34.89 | -0.143 | Cy |
| ZoGSTU16 | Maker00042725 | 216 | 24.58 | 5.14 | 35.78 | -0.118 | Cy |
| ZoGSTU17 | Maker00052435 | 237 | 25.65 | 5.67 | 23.52 | 0.175 | Cy |
| ZoGSTU18 | Maker00052130 | 202 | 22.36 | 7.78 | 39.56 | 0.117 | Cp |
| ZoGSTU19 | Maker00052070 | 236 | 25.51 | 5.30 | 30.69 | 0.262 | Cy |
| ZoGSTU20 | Maker00075976 | 313 | 35.78 | 4.55 | 48.14 | -0.332 | Cy |
| ZoGSTU21 | Maker00075948 | 237 | 26.04 | 4.86 | 32.24 | 0.078 | Cy |
| ZoGSTU22 | Maker00008319 | 223 | 25.38 | 7.71 | 40.26 | -0.146 | Cy |
| ZoGSTU23 | Maker00008163 | 110 | 12.21 | 9.51 | 46.97 | -0.215 | Cp |
| ZoGSTU24 | Maker00071927 | 233 | 26.10 | 5.37 | 36.13 | 0.203 | Cy |
| ZoGSTU25 | Maker00045210 | 233 | 26.42 | 5.34 | 24.14 | 0.042 | Cy |
| ZoGSTU26 | Maker00045476 | 232 | 25.95 | 5.35 | 32.96 | -0.001 | Cy |
| ZoGSTU27 | Maker00045093 | 233 | 26.43 | 5.92 | 25.92 | 0.012 | Cy |
| ZoGSTU28 | Maker00044883 | 233 | 26.32 | 6.11 | 27.64 | 0.045 | Cy |
表4 ZoGST蛋白质特征
Table 4 Protein characterization of ZoGSTs
| 名称 Name | 基因ID Gene ID | 长度/aa Length | 分子量/kD Mw | 等电点 pI | 不稳定指数 Instability index | 亲水性 Hydrophilicity | 亚细胞定位 Subcellular localization |
|---|---|---|---|---|---|---|---|
| ZoGSTDHAR1 | Maker00035113 | 330 | 37.20 | 5.06 | 51.08 | -0.278 | Cp,Cy,Mt |
| ZoGSTDHAR2 | Maker00052306 | 328 | 36.71 | 9.10 | 47.47 | -0.285 | Cp |
| ZoGSTDHAR3 | Maker00046027 | 350 | 39.34 | 8.79 | 44.65 | -0.139 | Mt |
| ZoGSTDHAR5 | Maker00035024 | 255 | 28.17 | 6.25 | 43.08 | -0.232 | Cp,Cy |
| ZoGSTDHAR4 | Maker00022334 | 267 | 29.40 | 6.91 | 48.36 | -0.098 | Cp,Cy |
| ZoGSTEF1G1 | Maker00033503 | 418 | 47.80 | 5.62 | 40.78 | -0.387 | Cy |
| ZoGSTEF1G2 | Maker00031875 | 419 | 47.96 | 5.68 | 39.85 | -0.346 | Cy |
| ZoGSTCHQ1 | Maker00046252 | 265 | 31.38 | 9.20 | 36.80 | -0.314 | Cy |
| ZoGSTT1 | Maker00029139 | 263 | 29.87 | 8.99 | 41.86 | -0.200 | Cy |
| ZoGSTL1 | Maker00033284 | 218 | 24.45 | 4.57 | 49.88 | -0.138 | Nu |
| ZoGSTL2 | Maker00059206 | 262 | 29.82 | 5.32 | 41.03 | -0.344 | Cp |
| ZoGSTL3 | Maker00009568 | 241 | 27.61 | 5.28 | 42.39 | -0.194 | Cy |
| ZoGSTZ1 | Maker00037272 | 346 | 39.86 | 6.47 | 34.33 | -0.469 | Mt |
| ZoGSTZ2 | Maker00013992 | 420 | 46.61 | 8.85 | 57.36 | -0.186 | Cy |
| ZoGSTZ3 | Maker00051190 | 490 | 55.36 | 4.71 | 53.11 | -0.735 | Cp |
| ZoGSTZ4 | Maker00021183 | 334 | 37.27 | 8.20 | 52.82 | -0.159 | Cp |
| ZoGSTZ5 | Maker00078047 | 402 | 44.92 | 5.87 | 44.01 | -0.161 | Cy |
| ZoGSTF1 | Maker00077732 | 675 | 72.51 | 5.62 | 60.97 | -0.818 | Nu |
| ZoGSTF2 | Maker00076791 | 220 | 24.34 | 6.91 | 55.02 | -0.311 | Cy |
| ZoGSTF3 | Maker00068242 | 318 | 35.47 | 6.56 | 54.65 | -0.470 | Cp,Cy,Nu |
| ZoGSTF4 | Maker00047494 | 221 | 25.42 | 7.76 | 50.63 | -0.422 | Cy |
| ZoGSTF5 | Maker00076803 | 221 | 25.31 | 6.97 | 55.37 | -0.513 | Cy |
| ZoGSTF6 | Maker00050934 | 243 | 27.74 | 6.34 | 39.92 | -0.216 | Cy |
| ZoGSTF7 | Maker00023234 | 224 | 25.10 | 5.35 | 41.26 | -0.144 | Cy |
| ZoGSTF8 | Maker00077970 | 251 | 28.38 | 6.72 | 38.38 | 0.051 | Cy |
| ZoGSTF9 | Maker00078922 | 217 | 24.39 | 5.52 | 49.34 | -0.089 | Cy |
| ZoGSTF10 | Maker00078286 | 217 | 24.47 | 5.71 | 37.72 | 0.043 | Cy |
| ZoGSTF11 | Maker00078094 | 255 | 28.60 | 6.31 | 39.33 | 0.025 | Cy |
| ZoGSTF12 | Maker00077516 | 219 | 24.50 | 5.53 | 36.69 | 0.050 | Cy |
| ZoGSTF13 | Maker00040344 | 217 | 23.76 | 5.97 | 18.75 | 0.107 | Cy |
| ZoGSTU1 | Maker00008855 | 222 | 24.81 | 5.39 | 39.79 | -0.027 | Cy |
| ZoGSTU2 | Maker00015777 | 244 | 27.20 | 5.02 | 38.10 | -0.195 | Cy |
| ZoGSTU3 | Maker00049005 | 247 | 27.51 | 5.40 | 34.94 | -0.146 | Cy |
| ZoGSTU4 | Maker00045495 | 232 | 26.80 | 6.20 | 55.74 | -0.338 | Cy |
| ZoGSTU5 | Maker00045023 | 228 | 26.25 | 5.47 | 48.11 | -0.321 | Cy |
| ZoGSTU6 | Maker00061695 | 227 | 25.65 | 5.49 | 55.78 | 0.027 | Cy |
| ZoGSTU7 | Maker00045310 | 222 | 24.36 | 5.43 | 24.27 | 0.064 | Cy |
| ZoGSTU8 | Maker00042846 | 222 | 24.37 | 5.43 | 23.19 | 0.060 | Cy |
| ZoGSTU9 | Maker00044620 | 219 | 24.93 | 5.29 | 34.73 | -0.096 | Cy |
| ZoGSTU10 | Maker00042772 | 219 | 24.91 | 5.39 | 34.62 | -0.086 | Cy |
| ZoGSTU11 | Maker00042767 | 117 | 13.53 | 5.26 | 31.22 | -0.222 | Cp |
| ZoGSTU12 | Maker00042721 | 252 | 28.66 | 5.40 | 37.67 | -0.124 | Cy |
| ZoGSTU13 | Maker00042760 | 216 | 24.66 | 5.15 | 34.00 | -0.111 | Cy |
| ZoGSTU14 | Maker00045562 | 252 | 28.79 | 5.28 | 38.35 | -0.142 | Cy |
| ZoGSTU15 | Maker00045539 | 216 | 24.59 | 5.25 | 34.89 | -0.143 | Cy |
| ZoGSTU16 | Maker00042725 | 216 | 24.58 | 5.14 | 35.78 | -0.118 | Cy |
| ZoGSTU17 | Maker00052435 | 237 | 25.65 | 5.67 | 23.52 | 0.175 | Cy |
| ZoGSTU18 | Maker00052130 | 202 | 22.36 | 7.78 | 39.56 | 0.117 | Cp |
| ZoGSTU19 | Maker00052070 | 236 | 25.51 | 5.30 | 30.69 | 0.262 | Cy |
| ZoGSTU20 | Maker00075976 | 313 | 35.78 | 4.55 | 48.14 | -0.332 | Cy |
| ZoGSTU21 | Maker00075948 | 237 | 26.04 | 4.86 | 32.24 | 0.078 | Cy |
| ZoGSTU22 | Maker00008319 | 223 | 25.38 | 7.71 | 40.26 | -0.146 | Cy |
| ZoGSTU23 | Maker00008163 | 110 | 12.21 | 9.51 | 46.97 | -0.215 | Cp |
| ZoGSTU24 | Maker00071927 | 233 | 26.10 | 5.37 | 36.13 | 0.203 | Cy |
| ZoGSTU25 | Maker00045210 | 233 | 26.42 | 5.34 | 24.14 | 0.042 | Cy |
| ZoGSTU26 | Maker00045476 | 232 | 25.95 | 5.35 | 32.96 | -0.001 | Cy |
| ZoGSTU27 | Maker00045093 | 233 | 26.43 | 5.92 | 25.92 | 0.012 | Cy |
| ZoGSTU28 | Maker00044883 | 233 | 26.32 | 6.11 | 27.64 | 0.045 | Cy |

图5 ZoGST在姜不同生长时期根茎(A)和生长90 d不同组织(B)中的表达
Fig. 5 Expression of ZoGST in rhizomes(A)of ginger at different growth stages and different tissues(B)growing for 90 days

图6 ZoGST在姜不同颜色组织(A)、根茎不同贮藏温度(B)及姜苗在褪黑素处理(C)下叶片中的表达
Fig. 6 Expression of ZoGST in ginger tissues of different colors(A),rhizomes at different storage temperatures(B)and leaves of ginger seedlings under melatonin treatment(C)

图7 ZoGST在姜不同土壤孔隙含水量下接种青枯菌(A)和根茎接种腐皮镰刀菌 + 壳聚糖处理(B)下的表达
Fig. 7 Expression of ZoGST under different soil pore moisture content of ginger inoculated with Ralstonia solanacearum(A)and rhizome inoculated with Fusarium solani + chitosan treatment(B)
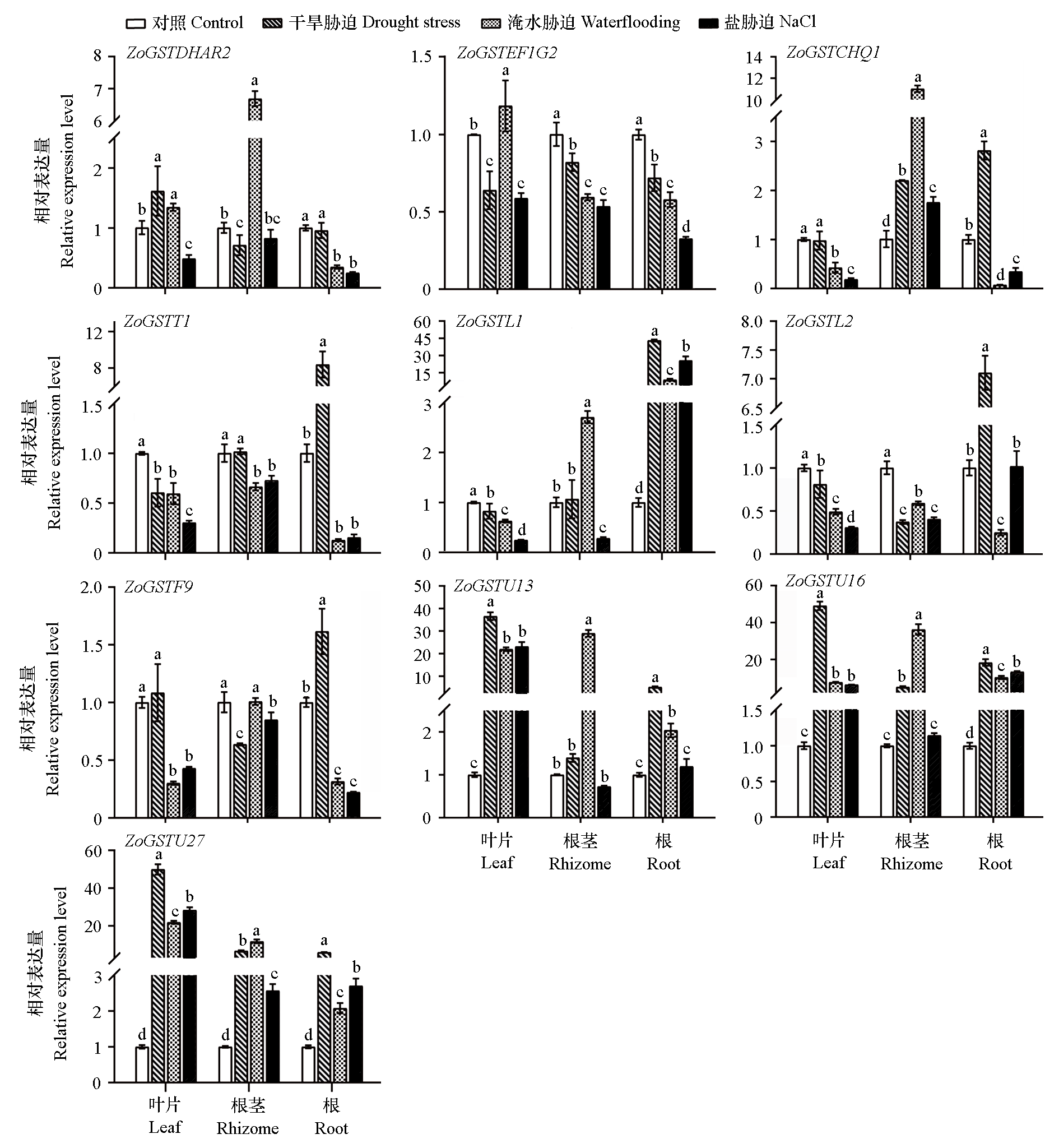
图8 ZoGST基因成员在不同胁迫下的基因表达特性 图柱上的不同字母代表相同组织不同处理间差异显著(P < 0.05)。
Fig. 8 Expression of ZoGST gene members under different stresses The different letters on the graph bar represent significant differences between different treatments of the same organization(P < 0.05).
| [1] |
|
| [2] |
|
| [3] |
|
|
毕艳, 郑伟, 周涛, 江维克, 杨昌贵, 肖承鸿, 周太敏. 2021. 太子参谷胱甘肽-S-转移酶基因家族生物信息学及表达分析. 广西植物, 41 (4):535-554.
|
|
| [4] |
doi: 10.16288/j.yczz.15-069 pmid: 26351171 |
|
曹运鹏, 方志, 李姝妹, 闫冲冲, 丁庆庆, 程曦, 林毅, 郭宁, 蔡永萍. 2015. 砀山酥梨4CL基因家族的全基因组鉴定与分析. 遗传, 37 (7):711-719.
pmid: 26351171 |
|
| [5] |
doi: 10.1093/dnares/dsq031 pmid: 21169340 |
| [6] |
|
| [7] |
doi: 10.1186/s12870-017-1179-z pmid: 29179697 |
| [8] |
|
| [9] |
|
| [10] |
doi: 10.1016/s1360-1385(00)01601-0 pmid: 10785664 |
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
|
姜栋柱, 赵小琴, 刘燃, 李洪雷, 刘奕清, 蔡小东. 2022. 姜种质资源农艺性状的遗传多样性分析. 中国蔬菜,(11):86-91.
|
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
|
李晓玉, 江海波, 江海洋, 朱苏文. 2013. 玉米全基因组谷胱苷肽-S-转移酶基因家族的分析. 安徽农业大学学报, 40 (3):350-356.
|
|
| [28] |
|
|
李永生, 方永丰, 李玥, 张同祯, 慕平, 王芳, 彭云玲, 王威, 张金文, 王汉宁. 2016. 玉米逆境响应基因ZmGST23克隆和表达分析. 农业生物技术学报, 24 (5):667-677.
|
|
| [29] |
|
|
李濯雪, 陈信波. 2015. 植物组织特异性启动子及相关顺式作用元件研究进展. 生物学杂志, 32 (6):91-95.
|
|
| [30] |
|
| [31] |
|
| [32] |
|
|
马凯恒, 谢牧洪, 郑欣悦, 贾彩霞, 洪心悦, 孙宇栋, 杨桂燕. 2021. 脱落酸和赤霉素对核桃JrGSTU8基因响应炭疽病的表达调控. 中南林业科技大学学报, 41 (12):121-129.
|
|
| [33] |
doi: 10.3724/SP.J.1006.2023.14207 |
|
马骊, 白静, 赵玉红, 孙柏林, 侯献飞, 方彦, 王旺田, 蒲媛媛, 刘丽君, 徐佳, 陶肖蕾, 孙万仓, 武军艳. 2023. 冷胁迫下甘蓝型冬油菜表达蛋白及BnGSTs基因家族的鉴定与分析. 作物学报, 49 (1):153-169.
doi: 10.3724/SP.J.1006.2023.14207 |
|
| [34] |
doi: 10.1104/pp.123.4.1561 pmid: 10938372 |
| [35] |
|
| [36] |
doi: 10.1111/j.1365-313X.2008.03761.x pmid: 19067976 |
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
|
王丽萍, 戚元成, 赵彦修, 张慧. 2002. 盐地碱蓬GST基因的克隆、序列分析及其表达特征. 植物生理与分子生物学学报,(2):133-136.
|
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
doi: 10.16288/j.yczz.16-435 pmid: 28903901 |
|
许磊, 陈文, 司国阳, 黄艺园, 林毅, 蔡永萍, 高俊山. 2017. 陆地棉GST基因家族全基因组分析. 遗传, 39 (8):737-752.
pmid: 28903901 |
|
| [47] |
|
|
张创娟, 程斌, 杨乐, 冷艳, 李师翁. 2022. 基于全基因组和转录组的绿豆谷胱甘肽转移酶基因及其对镉胁迫的响应. 西北农业学报, 31 (6):703-717.
|
|
| [48] |
|
| [49] |
|
| [50] |
pmid: 16622324 |
|
赵凤云, 王晓云, 赵彦修, 张慧. 2006. 转入盐地碱蓬谷胱甘肽转移酶和过氧化氢酶基因增强水稻幼苗对低温胁迫的抗性. 植物生理与分子生物学学报, 32 (2):231-238.
pmid: 16622324 |
|
| [51] |
doi: 10.7525/j.issn.1673-5102.2022.05.010 |
|
钟鑫爱, 孟诗琪, 周婉婷, 姚琦, 张琼, 兴旺, 刘大丽. 2022. 甜菜谷胱甘肽S-转移酶基因家族鉴定及在镉胁迫下的响应分析. 植物研究, 42 (5):790-801.
doi: 10.7525/j.issn.1673-5102.2022.05.010 |
| [1] | 郭昌权, 李丹琪, 惠馨冉, 郑婧雅, 侯梦璐, 朱永兴, . 姜DUF966基因家族成员鉴定与表达分析[J]. 园艺学报, 2024, 51(9): 2031-2047. |
| [2] | 赵佳莹, 曾周婷, 岑欣颖, 施姣淇, 李效贤, 沈晓霞, 俞振明, . 铁皮石斛CCO基因家族鉴定及其在花发育中的表达分析[J]. 园艺学报, 2024, 51(9): 2075-2088. |
| [3] | 陈佳悦, 段英明, 周雁, 肖扬, 边银丙, 龚钰华. 香菇ALDH家族基因鉴定、表达及功能分析[J]. 园艺学报, 2024, 51(5): 1033-1046. |
| [4] | 刘晋红, 王峥, 于昊, 辛依睿, 亓果宁, 柳参奎, 任慧敏. 毛竹SLAC家族基因鉴定及PheSLAC1功能分析[J]. 园艺学报, 2024, 51(3): 545-559. |
| [5] | 罗新锐, 张晓旭, 王玉萍, 王智, 马媛媛, 周丙月. 普通菜豆Trihelix基因家族鉴定及表达分析[J]. 园艺学报, 2024, 51(12): 2775-2790. |
| [6] | 王子妍 , 吴 燕 , 杨欣宇 , 许剑辉 , 丁 琼 , 方晋翔 , 章 起 , 汪伟民 , 陈友根 , . 姜新品种‘天白姜 1 号’[J]. 园艺学报, 2023, 50(S1): 39-40. |
| [7] | 苏赤连 , 汤 聪 , 林 玲 , 陈士壬 , 刘文艺 , 黎 浩 , 龙简妹 , 刘 念 , . 姜花新品种‘和煦’ [J]. 园艺学报, 2023, 50(S1): 123-124. |
| [8] | 汤 聪 , 张 斌 , 李 斌 , 林 玲 , 刘文艺 , 苏赤连 , 刘 念 , 叶琼香 , 吴郁璇 . 姜花属花卉新品种‘晨曦’[J]. 园艺学报, 2023, 50(S1): 125-126. |
| [9] | 陆静, 韦素云, 尹佟明, 陈赢男. Type-A细胞分裂素响应调节因子基因家族研究进展[J]. 园艺学报, 2023, 50(9): 1867-1888. |
| [10] | 黄治皓, 刘婷婷, 董旭杰, 严明理, 刘志祥, 曾超珍. 芥菜HMA家族基因鉴定及其在镉胁迫下的表达分析[J]. 园艺学报, 2023, 50(6): 1230-1242. |
| [11] | 张爱玲, 涂红艳, 肖望, 钟晓晴, 陆秋婵, 成丽萍, 林晓萍, 麦钰玲. 白姜花二倍体与四倍体切花形态与显微结构变化观察[J]. 园艺学报, 2023, 50(2): 345-358. |
| [12] | 于婷婷, 李欢, 宁源生, 宋建飞, 彭璐琳, 贾竣淇, 张玮玮, 杨洪强. 苹果GRAS全基因组鉴定及其对生长素的响应分析[J]. 园艺学报, 2023, 50(2): 397-409. |
| [13] | 申玉晓, 邹金玉, 罗平, 尚文倩, 李永华, 何松林, 王政, 石力匀. ‘月月粉’月季PP2C家族基因鉴定及非生物胁迫响应分析[J]. 园艺学报, 2023, 50(10): 2139-2156. |
| [14] | 翟含含, 翟宇杰, 田义, 张叶, 杨丽, 温陟良, 陈海江. 桃SAUR家族基因分析及PpSAUR5功能鉴定[J]. 园艺学报, 2023, 50(1): 1-14. |
| [15] | 包 苹, 蔡邦平, 高小坤, 谢雨萱, 蔡长福, 王梦园, 王金英, 丁友芳, 陈伯毅. 姜花新品种‘霓裳’[J]. 园艺学报, 2022, 49(S1): 141-142. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2012 《园艺学报》编辑部 京ICP备10030308号-2 国际联网备案号 11010802023439
编辑部地址: 北京市海淀区中关村南大街12号中国农业科学院蔬菜花卉研究所 邮编: 100081
电话: 010-82109523 E-Mail: yuanyixuebao@126.com
技术支持:北京玛格泰克科技发展有限公司