
园艺学报 ›› 2024, Vol. 51 ›› Issue (10): 2267-2280.doi: 10.16420/j.issn.0513-353x.2023-0723
张淑文1, 梁森苗1, 俞浙萍1, 孙鹂1, 叶海萍3, 胡晓金4, 颜丽菊6, 吴玉勇5, 应铮峥5, 郑锡良1, 戚行江1,2,*( )
)
收稿日期:2024-05-28
修回日期:2024-08-08
出版日期:2024-10-25
发布日期:2024-10-21
通讯作者:
基金资助:
ZHANG Shuwen1, LIANG Senmiao1, YU Zheping1, SUN Li1, YE Haiping3, HU Xiaojin4, YAN Liju6, WU Yuyong5, YING Zhengzheng5, ZHENG Xiliang1, QI Xingjiang1,2,*( )
)
Received:2024-05-28
Revised:2024-08-08
Published:2024-10-25
Online:2024-10-21
摘要:
基于全基因组重测序技术(whole genome sequencing,WGS),挖掘覆盖杨梅全基因组的SNP,明确杨梅基因组SNP的分布特点,并开展36份杨梅老树种质的群体遗传结构分析。由36份杨梅老树种质共获得高质量测序数据398.26 Gb,测序深度为37.34×,平均GC含量为38.57%,Q30为97.56%。共检测到5 362 434个SNP位点,1 ~ 8号染色体的SNP数量在655 299 ~ 897 575之间,共包含16个SNP变异惰性区域,总长度为9.45 Mb。邻接法进化树分析,36份种质可聚为3个群体,云南腾冲的6份种质聚为群体Ⅰ,与其他种质亲缘关系较远;湖南靖州的4份种质聚为群体Ⅱ;其余26份种质聚类为群体Ⅲ,其中浙江黄岩与仙居种质亲缘关系最近,以‘东魁’为主。群体连锁不平衡(LD)分析发现,云南腾冲种群LD衰减速率最快,浙江黄岩种群体次之,浙江慈溪种群最慢。对浙江黄岩(‘东魁’)、慈溪(‘荸荠种’)进行群体间遗传分化指数(Fst)分析,群体间差异显著的基因功能以抗逆性、次生代谢、植株形态建成等为主,进而形成这两个品种的表型差异。群体结构分析发现,福建、湖南靖州、湖北来凤等种群均包含了浙江仙居种质的遗传背景,且在湖南靖州种群中也发现了浙江仙居种群的杂交信号,表明近百年来,浙江仙居杨梅种质对福建、湖南、湖北等地栽培品种的形成影响较大。
张淑文, 梁森苗, 俞浙萍, 孙鹂, 叶海萍, 胡晓金, 颜丽菊, 吴玉勇, 应铮峥, 郑锡良, 戚行江. 杨梅老树种质群体遗传结构的WGS分析[J]. 园艺学报, 2024, 51(10): 2267-2280.
ZHANG Shuwen, LIANG Senmiao, YU Zheping, SUN Li, YE Haiping, HU Xiaojin, YAN Liju, WU Yuyong, YING Zhengzheng, ZHENG Xiliang, QI Xingjiang. Population Structure Analysis of Old Germplasms of Chinese Bayberry Based on WGS[J]. Acta Horticulturae Sinica, 2024, 51(10): 2267-2280.
| 采样地点 Sampling location | 经度Longitude | 纬度 Latitude | 海拔/m Altitude | 品种 Cultivar | 种质编号 Number | 估计树龄/a Tree-age(estimated) |
|---|---|---|---|---|---|---|
| 浙江黄岩 Huangyan,Zhejiang | 121.17° | 28.36° | 30 ~ 60 | 东魁 Dongkui | HY-1,HY-2,HY-4 | 70 |
| HY-5 | 180 | |||||
| HY-9 | 40 | |||||
| 浙江仙居 Xianju,Zhejiang | 120.47° | 28.76° | 150 ~ 170 | 水梅 Shuimei | XJ-1,XJ-3,XJ-5, XJ-7,XJ-9 | 100 |
| 浙江慈溪 Cixi,Zhejiang | 121.15° | 30.56° | 30 | 荸荠种 Biqizhong | CX-1,CX-2,CX-3,CX-4,CX-5 | 120 |
| 湖南靖州 Jingzhou,Hunan | 109.63° | 26.66° | 370 | 木洞大叶 Mudong Daye | HN-1 | 120 |
| HN-2 | 130 | |||||
| 上冲梅 Shangchongmei | HN-3,HN-7 | 110 | ||||
| HN-4 | 120 | |||||
| 湖北来凤 Laifeng,Hubei | 109.29° | 29.61° | 580 ~ 695 | 红梅 Hongmei | HB-1,HB-2 | 150 |
| HB-4,HB-5 | 100 | |||||
| 乌梅 Wumei | HB-6 | 100 | ||||
| 云南腾冲 Tengchong,Yunnan | 98.34° | 25.19° | 1 550 ~ 1 600 | 野生种质 Wild germplasm | YN-1,YN-6 | 190 |
| YN-2 | 130 | |||||
| YN-3,YN-4 | 200 | |||||
| YN-5 | 300 | |||||
| 浙江临海 (原产地福建) Linhai,Zhejiang(Origin is Fujian Province) | 119.28° | 26.08° | 130 | 硬丝 Yingsi | FJ-1 | 30 |
| 软丝 Ruansi | FJ-2 | |||||
| 浮宫一号Fugong 1 | FJ-3 | |||||
| 安海王子Anhai Wangzi | FJ-4 | |||||
| 安海早 Anhaizao | FJ-5 |
表1 杨梅老树种质资源信息
Table 1 The information of Chinese bayberry old germplasm resources
| 采样地点 Sampling location | 经度Longitude | 纬度 Latitude | 海拔/m Altitude | 品种 Cultivar | 种质编号 Number | 估计树龄/a Tree-age(estimated) |
|---|---|---|---|---|---|---|
| 浙江黄岩 Huangyan,Zhejiang | 121.17° | 28.36° | 30 ~ 60 | 东魁 Dongkui | HY-1,HY-2,HY-4 | 70 |
| HY-5 | 180 | |||||
| HY-9 | 40 | |||||
| 浙江仙居 Xianju,Zhejiang | 120.47° | 28.76° | 150 ~ 170 | 水梅 Shuimei | XJ-1,XJ-3,XJ-5, XJ-7,XJ-9 | 100 |
| 浙江慈溪 Cixi,Zhejiang | 121.15° | 30.56° | 30 | 荸荠种 Biqizhong | CX-1,CX-2,CX-3,CX-4,CX-5 | 120 |
| 湖南靖州 Jingzhou,Hunan | 109.63° | 26.66° | 370 | 木洞大叶 Mudong Daye | HN-1 | 120 |
| HN-2 | 130 | |||||
| 上冲梅 Shangchongmei | HN-3,HN-7 | 110 | ||||
| HN-4 | 120 | |||||
| 湖北来凤 Laifeng,Hubei | 109.29° | 29.61° | 580 ~ 695 | 红梅 Hongmei | HB-1,HB-2 | 150 |
| HB-4,HB-5 | 100 | |||||
| 乌梅 Wumei | HB-6 | 100 | ||||
| 云南腾冲 Tengchong,Yunnan | 98.34° | 25.19° | 1 550 ~ 1 600 | 野生种质 Wild germplasm | YN-1,YN-6 | 190 |
| YN-2 | 130 | |||||
| YN-3,YN-4 | 200 | |||||
| YN-5 | 300 | |||||
| 浙江临海 (原产地福建) Linhai,Zhejiang(Origin is Fujian Province) | 119.28° | 26.08° | 130 | 硬丝 Yingsi | FJ-1 | 30 |
| 软丝 Ruansi | FJ-2 | |||||
| 浮宫一号Fugong 1 | FJ-3 | |||||
| 安海王子Anhai Wangzi | FJ-4 | |||||
| 安海早 Anhaizao | FJ-5 |
| SNP位置SNP ID | F1 | F2 | R |
|---|---|---|---|
| Chr1:9855573 | TTGTCCACATCAATGCCGCAT | TTGTCCACATCAATGCCGCAC | GTTGGATTAGATACTTATGACATTGCTACG |
| Chr1:24875516 | AGGCACTCAGCCTTGCTGCTC | AGGCACTCAGCCTTGCTGCTG | GCGACTTAACTGTTATTATCACAGC |
| Chr2:1096506 | CTGAGTGTAGATAGAATAGAGCTCC | CTGAGTGTAGATAGAATAGAGCTCT | GGGATATGCACTTACGGATTGC |
| Chr2:34942573 | GCTATGTAAAAATACGTATCGCCAG | GCTATGTAAAAATACGTATCGCCAT | GGCAATCTCTACCAGCATAATG |
| Chr3:3035320 | TACTGCTTTTCTTTTATC | TACTGCTTTTCTTTTATT | TGAGCCCCAAAGATATTGCAAGAACATAAC |
| Chr3:25557841 | TCTAGGATGAGATTTTGTG | TCTAGGATGAGATTTTGTT | TTCAAAGACAAGAATGCAGCATG |
| Chr4:2150997 | GAAGGTGACCAAGTTCATGCTGTACTCAATTTTTGACTT | GAAGGTCGGAGTCAACGGATTGTACTCAATTTTTGACTC | TTCAAAGCCACATCCAACATCA |
| Chr4:30168356 | CTCATCACCAAGAGTCTTCCG | CTCATCACCAAGAGTCTTCCA | CAGTTGACACTGACAGAGCTGG |
| Chr5:6997703 | CAGGATATGATCTAGGAAGTTGTGC | CAGGATATGATCTAGGAAGTTGTGT | TGGCGCAATCACCCCAATTTTAACTGGA |
| Chr5:19892053 | GGTCGATTTGTGGAGGTTTTTCAGT | GGTCGATTTGTGGAGGTTTTTCAGG | AGGCCTCGATGACCACTTTGCCACCCAC |
| Chr6:4338019 | CTCTTTGCAAAGCTCGCTC | CTCTTTGCAAAGCTCGCTT | TTTCTCAATCATAAGAGCGGCCTCTT |
| Chr6:17174448 | TGTTGATGGACCATCCACGGAGTAA | TGTTGATGGACCATCCACGGAGTAC | TTAATTTGTTTTTTATTTATCACC |
| Chr6:20374169 | GTTAAGTCATCCCCTCAAT | GTTAAGTCATCCCCTCAAC | AAGCGACCTCTTCCTCACACAA |
| Chr6:28747343 | TGCTGAAATGAGGCTCAGTGG | TGCTGAAATGAGGCTCAGTGA | AATGATCAGGCTTCATAAGCTC |
| Chr6:32048517 | CCTCCTAGAATTTCAACGCTCTTAA | CCTCCTAGAATTTCAACGCTCTTAT | AACATGACTCGTTTGGTTCAAAT |
| Chr7:7268907 | GCAACAATCTTCCTCGCTTTT | GCAACAATCTTCCTCGCTTTC | GAAAGAGAAGCCCTCCTCAAATTCAGTCAG |
| Chr7:31486783 | CCTCGAAATGTGGAGGTCCCAGC | CCTCGAAATGTGGAGGTCCCAGT | CATGGCGCAATCACCAAATCCCATACTG |
| Chr8:7003270 | CCAACAAATGATAGCGCTTTAG | CCAACAAATGATAGCGCTTTAA | GCTAAAAGTCTTGAGGGTATTA |
| Chr8:18551161 | GGTTGTGAGAGGATTAAAAGTA | GGTTGTGAGAGGATTAAAAGTG | AAATTCCAATTGCATGAGGTTTTCGG |
| Chr8:28476059 | TTCTGCGTTCTCCGACGTTCTTCCC | TTCTGCGTTCTCCGACGTTCTTCCT | TGCATGTGTTATGAACGGATGT |
表2 KASP引物序列信息
Table 2 The KASP primer sequence
| SNP位置SNP ID | F1 | F2 | R |
|---|---|---|---|
| Chr1:9855573 | TTGTCCACATCAATGCCGCAT | TTGTCCACATCAATGCCGCAC | GTTGGATTAGATACTTATGACATTGCTACG |
| Chr1:24875516 | AGGCACTCAGCCTTGCTGCTC | AGGCACTCAGCCTTGCTGCTG | GCGACTTAACTGTTATTATCACAGC |
| Chr2:1096506 | CTGAGTGTAGATAGAATAGAGCTCC | CTGAGTGTAGATAGAATAGAGCTCT | GGGATATGCACTTACGGATTGC |
| Chr2:34942573 | GCTATGTAAAAATACGTATCGCCAG | GCTATGTAAAAATACGTATCGCCAT | GGCAATCTCTACCAGCATAATG |
| Chr3:3035320 | TACTGCTTTTCTTTTATC | TACTGCTTTTCTTTTATT | TGAGCCCCAAAGATATTGCAAGAACATAAC |
| Chr3:25557841 | TCTAGGATGAGATTTTGTG | TCTAGGATGAGATTTTGTT | TTCAAAGACAAGAATGCAGCATG |
| Chr4:2150997 | GAAGGTGACCAAGTTCATGCTGTACTCAATTTTTGACTT | GAAGGTCGGAGTCAACGGATTGTACTCAATTTTTGACTC | TTCAAAGCCACATCCAACATCA |
| Chr4:30168356 | CTCATCACCAAGAGTCTTCCG | CTCATCACCAAGAGTCTTCCA | CAGTTGACACTGACAGAGCTGG |
| Chr5:6997703 | CAGGATATGATCTAGGAAGTTGTGC | CAGGATATGATCTAGGAAGTTGTGT | TGGCGCAATCACCCCAATTTTAACTGGA |
| Chr5:19892053 | GGTCGATTTGTGGAGGTTTTTCAGT | GGTCGATTTGTGGAGGTTTTTCAGG | AGGCCTCGATGACCACTTTGCCACCCAC |
| Chr6:4338019 | CTCTTTGCAAAGCTCGCTC | CTCTTTGCAAAGCTCGCTT | TTTCTCAATCATAAGAGCGGCCTCTT |
| Chr6:17174448 | TGTTGATGGACCATCCACGGAGTAA | TGTTGATGGACCATCCACGGAGTAC | TTAATTTGTTTTTTATTTATCACC |
| Chr6:20374169 | GTTAAGTCATCCCCTCAAT | GTTAAGTCATCCCCTCAAC | AAGCGACCTCTTCCTCACACAA |
| Chr6:28747343 | TGCTGAAATGAGGCTCAGTGG | TGCTGAAATGAGGCTCAGTGA | AATGATCAGGCTTCATAAGCTC |
| Chr6:32048517 | CCTCCTAGAATTTCAACGCTCTTAA | CCTCCTAGAATTTCAACGCTCTTAT | AACATGACTCGTTTGGTTCAAAT |
| Chr7:7268907 | GCAACAATCTTCCTCGCTTTT | GCAACAATCTTCCTCGCTTTC | GAAAGAGAAGCCCTCCTCAAATTCAGTCAG |
| Chr7:31486783 | CCTCGAAATGTGGAGGTCCCAGC | CCTCGAAATGTGGAGGTCCCAGT | CATGGCGCAATCACCAAATCCCATACTG |
| Chr8:7003270 | CCAACAAATGATAGCGCTTTAG | CCAACAAATGATAGCGCTTTAA | GCTAAAAGTCTTGAGGGTATTA |
| Chr8:18551161 | GGTTGTGAGAGGATTAAAAGTA | GGTTGTGAGAGGATTAAAAGTG | AAATTCCAATTGCATGAGGTTTTCGG |
| Chr8:28476059 | TTCTGCGTTCTCCGACGTTCTTCCC | TTCTGCGTTCTCCGACGTTCTTCCT | TGCATGTGTTATGAACGGATGT |
| 种质编号 Sample code | 有效数据量/Gb Clean base | GC含量/% GC content | Q20/% | Q30/% | 重复率/% Duplication rate | 测序深度/× Average depth |
|---|---|---|---|---|---|---|
| HY-1 | 10.69 | 38.18 | 99.96 | 97.61 | 35.40 | 36.09 |
| HY-2 | 10.36 | 38.00 | 99.96 | 97.63 | 36.83 | 34.97 |
| HY-4 | 10.45 | 39.43 | 99.96 | 97.72 | 39.03 | 35.27 |
| HY-5 | 12.17 | 39.51 | 99.96 | 97.72 | 35.50 | 41.09 |
| HY-9 | 11.58 | 38.34 | 99.96 | 97.72 | 35.30 | 39.07 |
| XJ-1 | 11.38 | 38.09 | 99.89 | 97.53 | 35.40 | 38.42 |
| XJ-3 | 10.52 | 39.18 | 99.88 | 97.66 | 33.84 | 35.50 |
| XJ-5 | 10.75 | 39.07 | 99.88 | 97.63 | 36.80 | 36.29 |
| XJ-7 | 10.40 | 38.05 | 99.93 | 97.69 | 34.34 | 35.12 |
| XJ-9 | 11.72 | 38.43 | 99.94 | 97.57 | 36.30 | 39.55 |
| CX-1 | 10.32 | 38.16 | 99.92 | 97.52 | 29.90 | 34.84 |
| CX-2 | 10.99 | 38.08 | 99.93 | 97.66 | 36.70 | 37.11 |
| CX-3 | 11.11 | 38.24 | 99.93 | 97.51 | 35.91 | 37.51 |
| CX-4 | 10.60 | 38.00 | 99.95 | 97.58 | 34.70 | 35.77 |
| CX-5 | 10.24 | 39.14 | 99.95 | 97.70 | 30.24 | 34.57 |
| HN-1 | 12.86 | 39.00 | 99.97 | 97.62 | 37.40 | 43.40 |
| HN-2 | 11.12 | 39.13 | 99.96 | 97.64 | 34.70 | 37.54 |
| HN-3 | 11.20 | 39.11 | 99.96 | 97.62 | 38.10 | 37.81 |
| HN-4 | 10.06 | 38.00 | 99.96 | 97.66 | 35.30 | 33.96 |
| HN-7 | 10.90 | 39.24 | 99.96 | 97.62 | 36.27 | 36.80 |
| HB-1 | 10.22 | 40.04 | 99.93 | 97.68 | 35.20 | 34.48 |
| HB-2 | 11.88 | 39.00 | 99.94 | 97.56 | 38.44 | 40.11 |
| HB-4 | 11.14 | 40.30 | 99.94 | 97.64 | 36.80 | 37.62 |
| HB-5 | 10.75 | 38.00 | 99.96 | 97.79 | 36.24 | 36.30 |
| HB-6 | 11.42 | 38.27 | 99.96 | 97.59 | 35.15 | 38.54 |
| FJ-1 | 10.01 | 38.14 | 99.99 | 97.00 | 27.05 | 33.80 |
| FJ-2 | 16.08 | 38.42 | 99.99 | 97.62 | 27.70 | 54.28 |
| FJ-3 | 10.35 | 38.00 | 99.99 | 96.94 | 25.20 | 34.93 |
| FJ-4 | 11.59 | 38.28 | 99.99 | 97.06 | 25.33 | 39.13 |
| FJ-5 | 11.75 | 38.12 | 99.99 | 97.63 | 25.42 | 39.68 |
| YN-1 | 10.63 | 38.51 | 99.93 | 97.72 | 30.77 | 35.88 |
| YN-2 | 10.81 | 38.34 | 99.93 | 97.44 | 31.30 | 36.51 |
| YN-3 | 10.78 | 38.07 | 99.93 | 97.46 | 31.04 | 36.40 |
| YN-4 | 10.54 | 38.16 | 99.93 | 97.48 | 29.22 | 35.57 |
| YN-5 | 10.50 | 38.31 | 99.93 | 97.36 | 30.62 | 35.46 |
| YN-6 | 10.37 | 38.16 | 99.93 | 97.49 | 28.15 | 35.01 |
| 平均Average | 11.06 | 38.57 | 99.95 | 97.56 | 33.38 | 37.34 |
表3 样本测序信息
Table 3 Sample re-sequencing information
| 种质编号 Sample code | 有效数据量/Gb Clean base | GC含量/% GC content | Q20/% | Q30/% | 重复率/% Duplication rate | 测序深度/× Average depth |
|---|---|---|---|---|---|---|
| HY-1 | 10.69 | 38.18 | 99.96 | 97.61 | 35.40 | 36.09 |
| HY-2 | 10.36 | 38.00 | 99.96 | 97.63 | 36.83 | 34.97 |
| HY-4 | 10.45 | 39.43 | 99.96 | 97.72 | 39.03 | 35.27 |
| HY-5 | 12.17 | 39.51 | 99.96 | 97.72 | 35.50 | 41.09 |
| HY-9 | 11.58 | 38.34 | 99.96 | 97.72 | 35.30 | 39.07 |
| XJ-1 | 11.38 | 38.09 | 99.89 | 97.53 | 35.40 | 38.42 |
| XJ-3 | 10.52 | 39.18 | 99.88 | 97.66 | 33.84 | 35.50 |
| XJ-5 | 10.75 | 39.07 | 99.88 | 97.63 | 36.80 | 36.29 |
| XJ-7 | 10.40 | 38.05 | 99.93 | 97.69 | 34.34 | 35.12 |
| XJ-9 | 11.72 | 38.43 | 99.94 | 97.57 | 36.30 | 39.55 |
| CX-1 | 10.32 | 38.16 | 99.92 | 97.52 | 29.90 | 34.84 |
| CX-2 | 10.99 | 38.08 | 99.93 | 97.66 | 36.70 | 37.11 |
| CX-3 | 11.11 | 38.24 | 99.93 | 97.51 | 35.91 | 37.51 |
| CX-4 | 10.60 | 38.00 | 99.95 | 97.58 | 34.70 | 35.77 |
| CX-5 | 10.24 | 39.14 | 99.95 | 97.70 | 30.24 | 34.57 |
| HN-1 | 12.86 | 39.00 | 99.97 | 97.62 | 37.40 | 43.40 |
| HN-2 | 11.12 | 39.13 | 99.96 | 97.64 | 34.70 | 37.54 |
| HN-3 | 11.20 | 39.11 | 99.96 | 97.62 | 38.10 | 37.81 |
| HN-4 | 10.06 | 38.00 | 99.96 | 97.66 | 35.30 | 33.96 |
| HN-7 | 10.90 | 39.24 | 99.96 | 97.62 | 36.27 | 36.80 |
| HB-1 | 10.22 | 40.04 | 99.93 | 97.68 | 35.20 | 34.48 |
| HB-2 | 11.88 | 39.00 | 99.94 | 97.56 | 38.44 | 40.11 |
| HB-4 | 11.14 | 40.30 | 99.94 | 97.64 | 36.80 | 37.62 |
| HB-5 | 10.75 | 38.00 | 99.96 | 97.79 | 36.24 | 36.30 |
| HB-6 | 11.42 | 38.27 | 99.96 | 97.59 | 35.15 | 38.54 |
| FJ-1 | 10.01 | 38.14 | 99.99 | 97.00 | 27.05 | 33.80 |
| FJ-2 | 16.08 | 38.42 | 99.99 | 97.62 | 27.70 | 54.28 |
| FJ-3 | 10.35 | 38.00 | 99.99 | 96.94 | 25.20 | 34.93 |
| FJ-4 | 11.59 | 38.28 | 99.99 | 97.06 | 25.33 | 39.13 |
| FJ-5 | 11.75 | 38.12 | 99.99 | 97.63 | 25.42 | 39.68 |
| YN-1 | 10.63 | 38.51 | 99.93 | 97.72 | 30.77 | 35.88 |
| YN-2 | 10.81 | 38.34 | 99.93 | 97.44 | 31.30 | 36.51 |
| YN-3 | 10.78 | 38.07 | 99.93 | 97.46 | 31.04 | 36.40 |
| YN-4 | 10.54 | 38.16 | 99.93 | 97.48 | 29.22 | 35.57 |
| YN-5 | 10.50 | 38.31 | 99.93 | 97.36 | 30.62 | 35.46 |
| YN-6 | 10.37 | 38.16 | 99.93 | 97.49 | 28.15 | 35.01 |
| 平均Average | 11.06 | 38.57 | 99.95 | 97.56 | 33.38 | 37.34 |
| 样本 Sample | SNP数量 Number of SNPs | 转换位点 Number of transition | 颠换位点 Number of transversion | 转换/颠换 Transition/ transversion | 杂合变异位点 Number of heterozygosity | 纯合变异位点 Number of homozygosity | 杂合率/% Heterozygosity rate |
|---|---|---|---|---|---|---|---|
| HY-1 | 2 050 938 | 1 411 884 | 639 054 | 2.21 | 1 724 369 | 326 569 | 84.08 |
| HY-2 | 2 048 642 | 1 410 307 | 638 335 | 2.21 | 1 721 860 | 326 782 | 84.05 |
| HY-4 | 2 052 202 | 1 412 718 | 639 484 | 2.21 | 1 724 844 | 327 358 | 84.05 |
| HY-5 | 2 052 967 | 1 413 221 | 639 746 | 2.21 | 1 723 585 | 329 382 | 83.96 |
| HY-9 | 2 052 974 | 1 413 453 | 639 521 | 2.21 | 1 725 777 | 327 197 | 84.06 |
| XJ-1 | 2 020 543 | 1 392 832 | 627 711 | 2.22 | 1 794 440 | 226 103 | 88.81 |
| XJ-3 | 2 015 957 | 1 389 488 | 626 469 | 2.22 | 1 788 704 | 227 253 | 88.73 |
| XJ-5 | 2 014 444 | 1 388 425 | 626 019 | 2.22 | 1 785 481 | 228 963 | 88.63 |
| XJ-7 | 2 019 041 | 1 391 447 | 627 594 | 2.22 | 1 792 393 | 226 648 | 88.77 |
| XJ-9 | 2 053 334 | 1 413 369 | 639 965 | 2.21 | 1 726 162 | 327 172 | 84.07 |
| CX-1 | 1 726 871 | 1 187 340 | 539 531 | 2.20 | 1 563 406 | 163 465 | 90.53 |
| CX-2 | 2 355 419 | 1 621 539 | 733 880 | 2.21 | 2 094 979 | 260 440 | 88.94 |
| CX-3 | 1 726 465 | 1 187 243 | 539 222 | 2.20 | 1 562 720 | 163 745 | 90.52 |
| CX-4 | 1 730 185 | 1 189 641 | 540 544 | 2.20 | 1 565 862 | 164 323 | 90.50 |
| CX-5 | 1 726 906 | 1 187 304 | 539 602 | 2.20 | 1 561 793 | 165 113 | 90.44 |
| HN-1 | 2 001 119 | 1 380 042 | 621 077 | 2.22 | 1 513 540 | 487 579 | 75.63 |
| HN-2 | 2 159 194 | 1 494 361 | 664 833 | 2.25 | 1 779 964 | 379 230 | 82.44 |
| HN-3 | 2 063 061 | 1 423 165 | 639 896 | 2.22 | 1 570 871 | 492 190 | 76.14 |
| HN-4 | 2 063 833 | 1 423 804 | 640 029 | 2.22 | 1 574 460 | 489 373 | 76.29 |
| HN-7 | 2 062 759 | 1 422 958 | 639 801 | 2.22 | 1 571 669 | 491 090 | 76.19 |
| HB-1 | 2 023 343 | 1 399 019 | 624 324 | 2.24 | 1 519 121 | 504 222 | 75.08 |
| HB-2 | 2 022 311 | 1 394 607 | 627 704 | 2.22 | 1 476 575 | 545 736 | 73.01 |
| HB-4 | 2 014 613 | 1 389 561 | 625 052 | 2.22 | 1 466 369 | 548 244 | 72.79 |
| HB-5 | 2 058 455 | 1 421 689 | 636 766 | 2.23 | 1 566 655 | 491 800 | 76.11 |
| HB-6 | 1 893 434 | 1 305 944 | 587 490 | 2.22 | 1 222 637 | 670 797 | 64.57 |
| FJ-1 | 2 049 201 | 1 417 215 | 631 986 | 2.24 | 1 636 785 | 412 416 | 79.87 |
| FJ-2 | 2 135 600 | 1 477 909 | 657 691 | 2.25 | 1 751 915 | 383 685 | 82.03 |
| FJ-3 | 2 059 344 | 1 422 225 | 637 119 | 2.23 | 1 645 218 | 414 126 | 79.89 |
| FJ-4 | 2 144 618 | 1 486 049 | 658 569 | 2.26 | 1 738 952 | 405 666 | 81.08 |
| FJ-5 | 2 109 931 | 1 460 493 | 649 438 | 2.25 | 1 725 461 | 384 470 | 81.78 |
| YN-1 | 3 517 942 | 2 430 232 | 1 087 710 | 2.23 | 966 536 | 2 551 406 | 27.47 |
| YN-2 | 3 508 535 | 2 422 505 | 1 086 030 | 2.23 | 913 854 | 2 594 681 | 26.05 |
| YN-3 | 3 512 213 | 2 426 292 | 1 085 921 | 2.23 | 952 693 | 2 559 520 | 27.13 |
| YN-4 | 3 505 810 | 2 421 699 | 1 084 111 | 2.23 | 943 055 | 2 562 755 | 26.90 |
| YN-5 | 3 499 575 | 2 416 870 | 1 082 705 | 2.23 | 884 303 | 2 615 272 | 25.27 |
| YN-6 | 3 504 092 | 2 418 865 | 1 085 227 | 2.23 | 857 631 | 2 646 461 | 24.48 |
表4 样本中检测到的SNP信息
Table 4 The SNP information detected in the 36 samples
| 样本 Sample | SNP数量 Number of SNPs | 转换位点 Number of transition | 颠换位点 Number of transversion | 转换/颠换 Transition/ transversion | 杂合变异位点 Number of heterozygosity | 纯合变异位点 Number of homozygosity | 杂合率/% Heterozygosity rate |
|---|---|---|---|---|---|---|---|
| HY-1 | 2 050 938 | 1 411 884 | 639 054 | 2.21 | 1 724 369 | 326 569 | 84.08 |
| HY-2 | 2 048 642 | 1 410 307 | 638 335 | 2.21 | 1 721 860 | 326 782 | 84.05 |
| HY-4 | 2 052 202 | 1 412 718 | 639 484 | 2.21 | 1 724 844 | 327 358 | 84.05 |
| HY-5 | 2 052 967 | 1 413 221 | 639 746 | 2.21 | 1 723 585 | 329 382 | 83.96 |
| HY-9 | 2 052 974 | 1 413 453 | 639 521 | 2.21 | 1 725 777 | 327 197 | 84.06 |
| XJ-1 | 2 020 543 | 1 392 832 | 627 711 | 2.22 | 1 794 440 | 226 103 | 88.81 |
| XJ-3 | 2 015 957 | 1 389 488 | 626 469 | 2.22 | 1 788 704 | 227 253 | 88.73 |
| XJ-5 | 2 014 444 | 1 388 425 | 626 019 | 2.22 | 1 785 481 | 228 963 | 88.63 |
| XJ-7 | 2 019 041 | 1 391 447 | 627 594 | 2.22 | 1 792 393 | 226 648 | 88.77 |
| XJ-9 | 2 053 334 | 1 413 369 | 639 965 | 2.21 | 1 726 162 | 327 172 | 84.07 |
| CX-1 | 1 726 871 | 1 187 340 | 539 531 | 2.20 | 1 563 406 | 163 465 | 90.53 |
| CX-2 | 2 355 419 | 1 621 539 | 733 880 | 2.21 | 2 094 979 | 260 440 | 88.94 |
| CX-3 | 1 726 465 | 1 187 243 | 539 222 | 2.20 | 1 562 720 | 163 745 | 90.52 |
| CX-4 | 1 730 185 | 1 189 641 | 540 544 | 2.20 | 1 565 862 | 164 323 | 90.50 |
| CX-5 | 1 726 906 | 1 187 304 | 539 602 | 2.20 | 1 561 793 | 165 113 | 90.44 |
| HN-1 | 2 001 119 | 1 380 042 | 621 077 | 2.22 | 1 513 540 | 487 579 | 75.63 |
| HN-2 | 2 159 194 | 1 494 361 | 664 833 | 2.25 | 1 779 964 | 379 230 | 82.44 |
| HN-3 | 2 063 061 | 1 423 165 | 639 896 | 2.22 | 1 570 871 | 492 190 | 76.14 |
| HN-4 | 2 063 833 | 1 423 804 | 640 029 | 2.22 | 1 574 460 | 489 373 | 76.29 |
| HN-7 | 2 062 759 | 1 422 958 | 639 801 | 2.22 | 1 571 669 | 491 090 | 76.19 |
| HB-1 | 2 023 343 | 1 399 019 | 624 324 | 2.24 | 1 519 121 | 504 222 | 75.08 |
| HB-2 | 2 022 311 | 1 394 607 | 627 704 | 2.22 | 1 476 575 | 545 736 | 73.01 |
| HB-4 | 2 014 613 | 1 389 561 | 625 052 | 2.22 | 1 466 369 | 548 244 | 72.79 |
| HB-5 | 2 058 455 | 1 421 689 | 636 766 | 2.23 | 1 566 655 | 491 800 | 76.11 |
| HB-6 | 1 893 434 | 1 305 944 | 587 490 | 2.22 | 1 222 637 | 670 797 | 64.57 |
| FJ-1 | 2 049 201 | 1 417 215 | 631 986 | 2.24 | 1 636 785 | 412 416 | 79.87 |
| FJ-2 | 2 135 600 | 1 477 909 | 657 691 | 2.25 | 1 751 915 | 383 685 | 82.03 |
| FJ-3 | 2 059 344 | 1 422 225 | 637 119 | 2.23 | 1 645 218 | 414 126 | 79.89 |
| FJ-4 | 2 144 618 | 1 486 049 | 658 569 | 2.26 | 1 738 952 | 405 666 | 81.08 |
| FJ-5 | 2 109 931 | 1 460 493 | 649 438 | 2.25 | 1 725 461 | 384 470 | 81.78 |
| YN-1 | 3 517 942 | 2 430 232 | 1 087 710 | 2.23 | 966 536 | 2 551 406 | 27.47 |
| YN-2 | 3 508 535 | 2 422 505 | 1 086 030 | 2.23 | 913 854 | 2 594 681 | 26.05 |
| YN-3 | 3 512 213 | 2 426 292 | 1 085 921 | 2.23 | 952 693 | 2 559 520 | 27.13 |
| YN-4 | 3 505 810 | 2 421 699 | 1 084 111 | 2.23 | 943 055 | 2 562 755 | 26.90 |
| YN-5 | 3 499 575 | 2 416 870 | 1 082 705 | 2.23 | 884 303 | 2 615 272 | 25.27 |
| YN-6 | 3 504 092 | 2 418 865 | 1 085 227 | 2.23 | 857 631 | 2 646 461 | 24.48 |
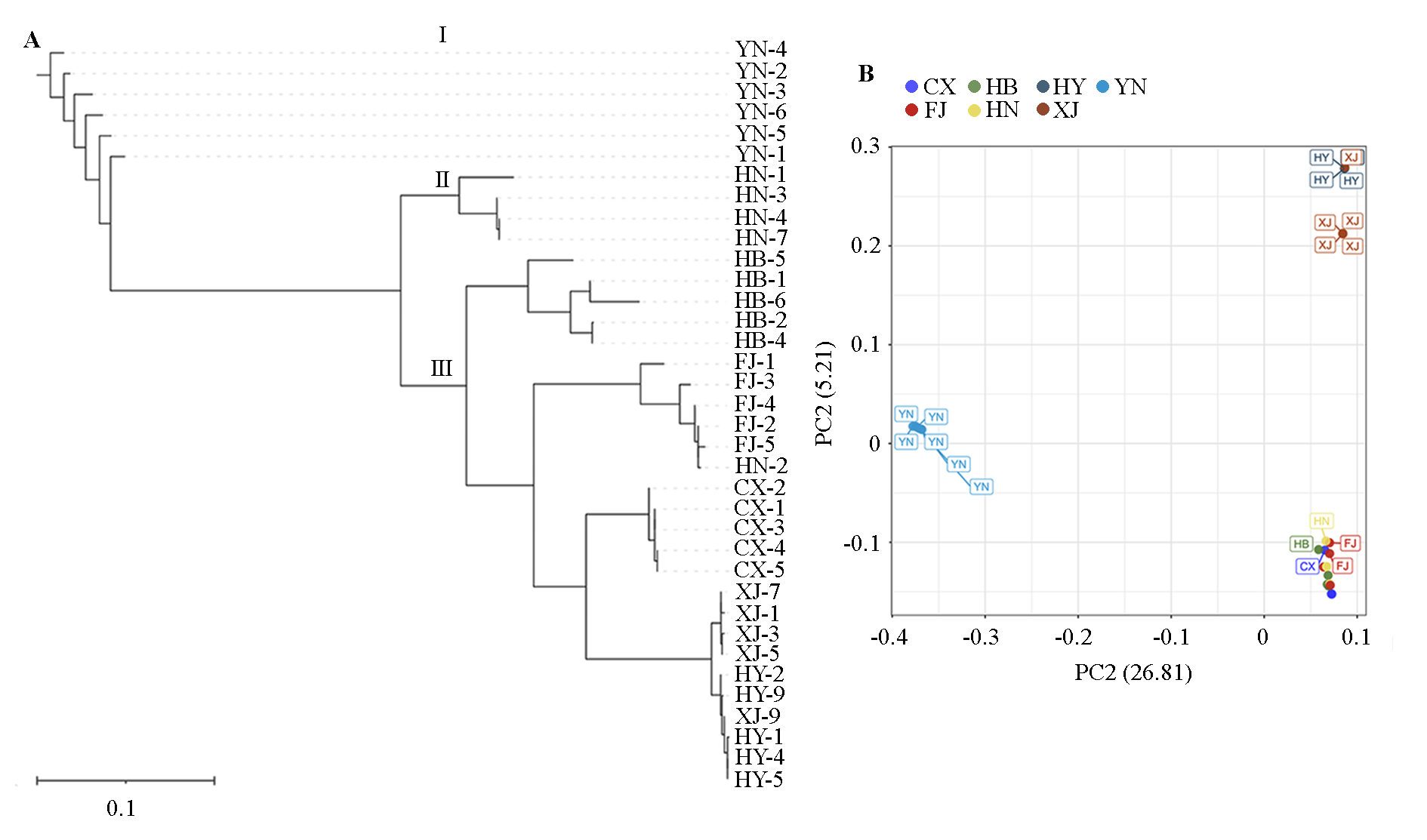
图2 杨梅老树种质群体进化树(A)与PCA分析(B) 36个种质详见表1。FJ:福建;CX:浙江慈溪;HB:湖北来凤;HN:湖南靖州;XJ:浙江仙居;HY:浙江黄岩;YN:云南腾冲。
Fig. 2 Population evolution tree(A)and PCA analysis(B)of old germplasms of Chinese bayberry The information of 36 germplasms was detailed in Table 1. FJ:Fujian;CX:Cixi,Zhejiang;HB:Laifeng,Hubei;HN:Jingzhou,Hunan;XJ:Xianju,Zhejiang;HY:Huangyan,Zhejiang;YN:Tengchong,Yunnan.
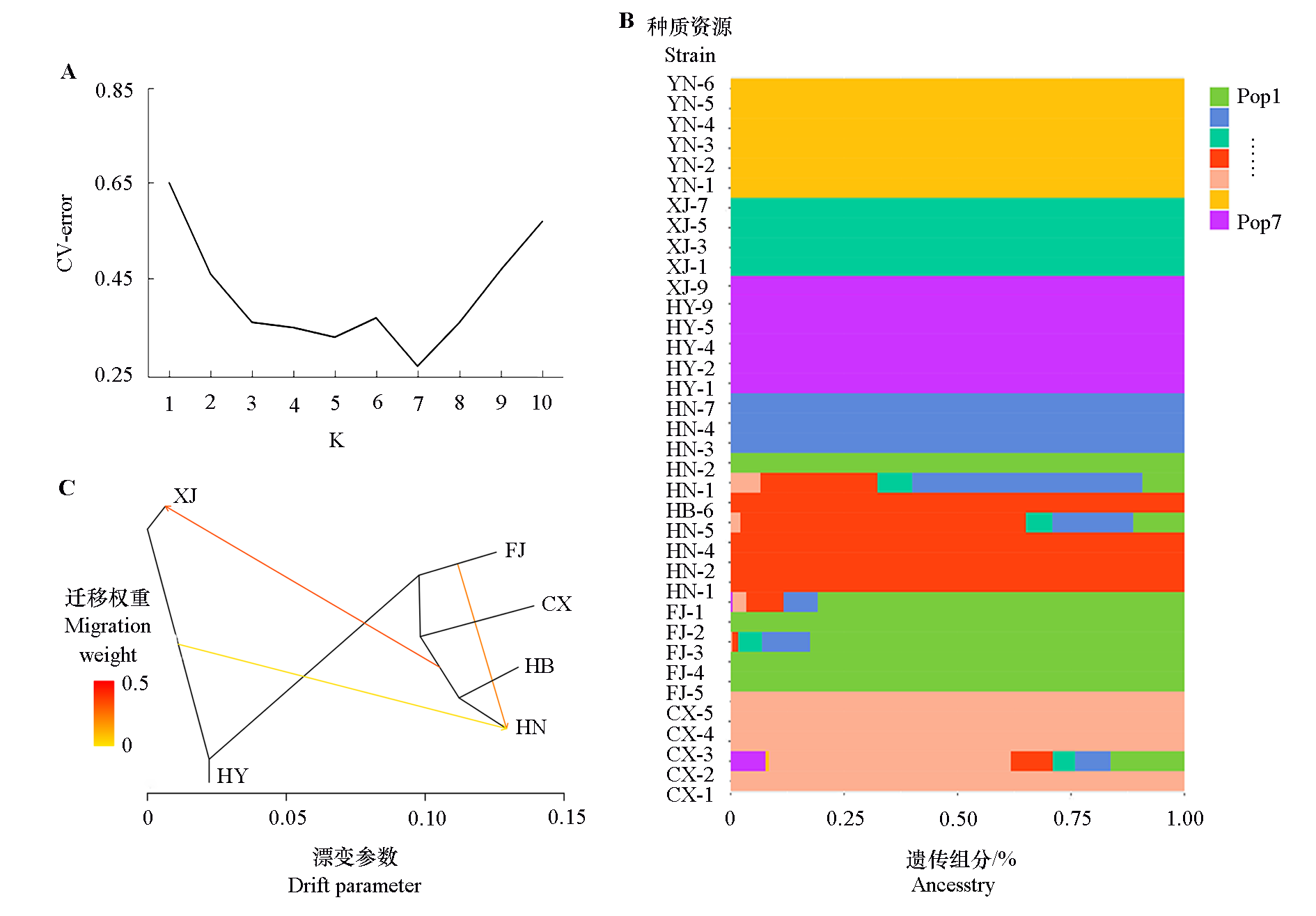
图3 K值的交叉验证错误率(A)、群体结构关系(B)和不同群体间的渗入信号(C) 36个种质详见表1。FJ:福建;CX:浙江慈溪;HB:湖北来凤;HN:湖南靖州;XJ:浙江仙居;HY:浙江黄岩;YN:云南腾冲。
Fig. 3 Cross validation(CV)error rate of K(A),population structure relationship(B)and signals of introgression among different populations(C) The information of 36 germplasms was detailed in Table 1. FJ:Fujian;CX:Cixi,Zhejiang;HB:Laifeng,Hubei;HN:Jingzhou,Hunan;XJ:Xianju,Zhejiang;HY:Huangyan,Zhejiang;YN:Tengchong,Yunnan.

图4 杨梅老树种质群体LD衰减速率(A)及浙江黄岩、慈溪群体间Fst分析(B) 36个种质详见表1。FJ:福建;CX:浙江慈溪;HB:湖北来凤;HN:湖南靖州;XJ:浙江仙居;HY:浙江黄岩;YN:云南腾冲。
Fig. 4 LD attenuation(A)and Fst analysis(B)between Huangyan and Cixi old germplasm population of Zhejiang The information of 36 germplasms was detailed in Table 1. FJ:Fujian;CX:Cixi,Zhejiang;HB:Laifeng,Hubei;HN:Jingzhou,Hunan;XJ:Xianju,Zhejiang;HY:Huangyan,Zhejiang;YN:Tengchong,Yunnan.
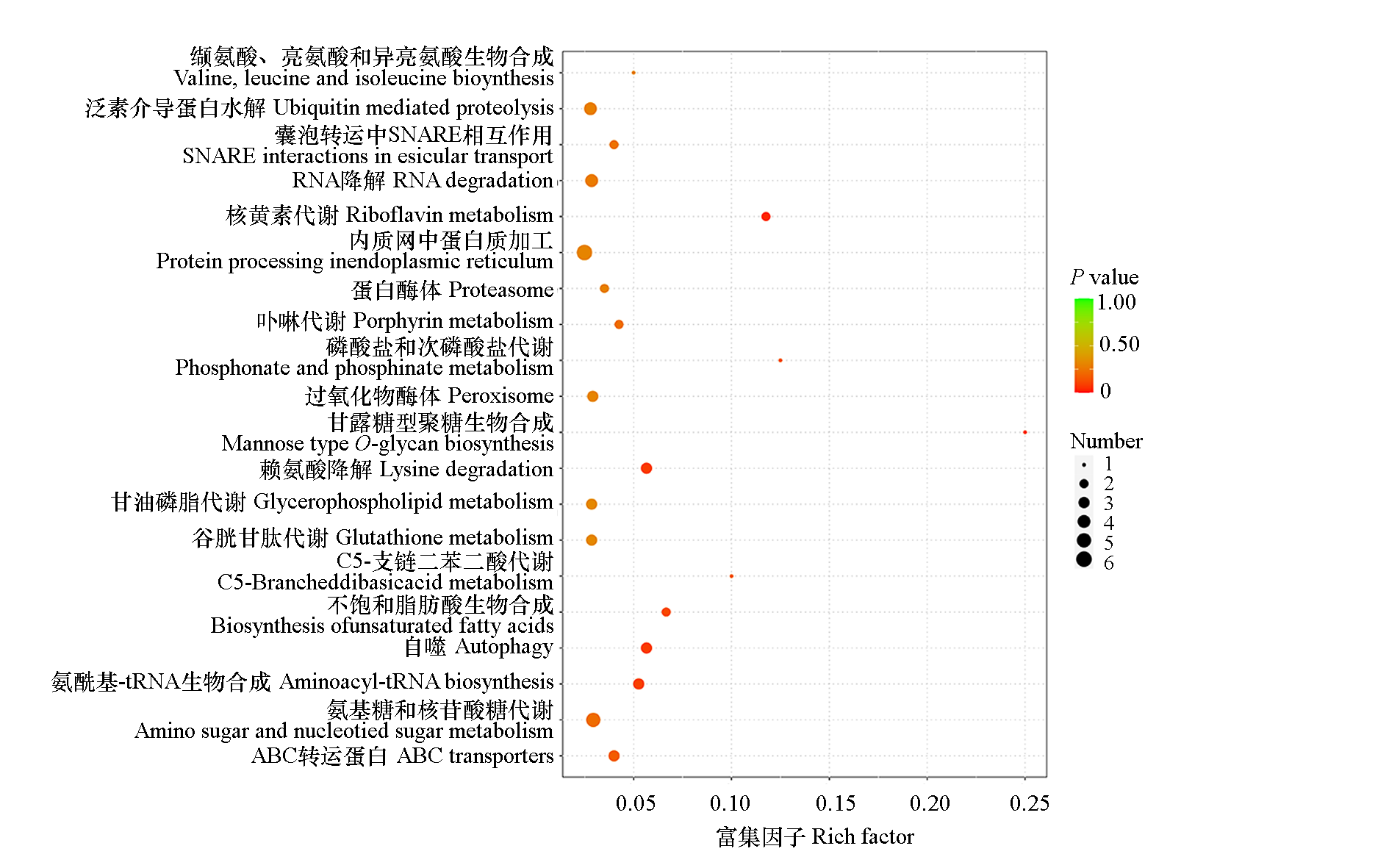
图6 杨梅‘东魁’和‘荸荠种’种质群体Fst前1%差异基因的KEGG富集通路分析
Fig. 6 KEGG enrichment pathway analysis of the top 1% differentially genes in the Fst of‘Dongkui’and‘Biqizhong’populations
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
doi: 10.1093/bioinformatics/btr330 pmid: 21653522 |
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
doi: 10.16420/j.issn.0513-353x.2022-0521 |
|
侯炳豪, 高婷, 魏月德, 高水练, 蔡银笔, 陈志明, 金珊, 张兴坦, 王鹏杰, 叶乃兴. 2023. 基于高深度基因组重测序的‘铁观音’茶树无性繁殖后代遗传变异研究. 园艺学报, 50 (7):1505-1517.
|
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
doi: 10.1093/bioinformatics/btp352 pmid: 19505943 |
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
|
张瑞, 张夏燚, 赵婷, 王双成, 张仲兴, 刘博, 张德, 王延秀. 2022. 基于转录组分析垂丝海棠响应盐碱胁迫的分子机制. 园艺学报, 49 (2):237-251.
doi: 10.16420/j.issn.0513-353x.2021-0077 |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
doi: 10.16420/j.issn.0513-353x.2018-0233 |
|
张淑文, 梁森苗, 郑锡良, 任海英, 朱婷婷, 戚行江. 2019. 杨梅基因组SSR引物的开发与应用. 园艺学报, 46 (1):149-156.
doi: 10.16420/j.issn.0513-353x.2018-0233 |
|
| [35] |
|
|
张淑文, 俞浙萍, 孙鹂, 武祥琪, 梁森苗, 郑锡良, 戚行江. 2022. 基于重测序的杨梅InDel标记开发与果实性状关联分析. 分子植物育种, 20 (6):1890-1900.
|
| [1] | 田雯, 李子琛, 王霖, 王大江, 王昆, 孙思邈, 王广艺, 刘昭, 路翔, 冯建荣, 高源. 苹果重要性状全基因组关联分析研究进展[J]. 园艺学报, 2024, 51(7): 1565-1579. |
| [2] | 秦宇, 郭荣琨, 农惠兰, 王燕, 崔凯, 董宁光. 利用SSR荧光标记构建山楂种质分子身份证[J]. 园艺学报, 2024, 51(6): 1227-1240. |
| [3] | 张文静, 徐大勇, 吴倩琳, 杨佛, 信丙越, 曾昕, 李峰. 拮抗番茄灰霉病的贝莱斯芽孢杆菌XDY66基因组分析[J]. 园艺学报, 2024, 51(6): 1413-1425. |
| [4] | 武祥琪, 孙鹂, 俞浙萍, 俞沁佩, 梁森苗, 郑锡良, 戚行江, 张淑文. 杨梅MrSPL4在响应干旱和低温胁迫中的作用研究[J]. 园艺学报, 2024, 51(5): 927-938. |
| [5] | 李品, 甘宁, 陈家伟, 项思翔, 沈静漪, 欧阳波, 卢永恩. 番茄自然群体磷利用效率分析及耐低磷种质筛选[J]. 园艺学报, 2024, 51(5): 993-1004. |
| [6] | 陈佳悦, 段英明, 周雁, 肖扬, 边银丙, 龚钰华. 香菇ALDH家族基因鉴定、表达及功能分析[J]. 园艺学报, 2024, 51(5): 1033-1046. |
| [7] | 蒋爽, 王晓庆, 施春晖, 李水根, 骆军. 利用PCR产物混池重测序检测梨褐皮关联SLAF 标签多态性[J]. 园艺学报, 2024, 51(4): 707-714. |
| [8] | 张正海, 王永富, 吴华茂, 于海龙, 曹亚从, 冯锡刚, 王立浩. 辣椒重要性状遗传定位与候选基因研究进展[J]. 园艺学报, 2024, 51(3): 669-696. |
| [9] | 董舒超, 洪骏, 凌嘉怡, 谢紫欣, 张胜军, 赵丽萍, 宋刘霞, 王银磊, 赵统敏. 番茄抗旱性的全基因组关联分析[J]. 园艺学报, 2024, 51(2): 229-238. |
| [10] | 徐琴, 王嘉颖, 张曼楠, 萧志浩, 郑涵楷, 卢永恩, 王涛涛, 张余洋, 张俊红, 叶志彪, 叶杰. 番茄苗期耐盐相关遗传位点鉴定及分子标记开发[J]. 园艺学报, 2024, 51(2): 239-252. |
| [11] | 袁青云, 韩昱, 贺巍, 苏会, 班秋艳, 吴春来, 周琼琼, 徐文静, 王丽鸳, 张芬. 茶树CsNPF6.1/6.3基因克隆及ABA转运功能验证[J]. 园艺学报, 2024, 51(12): 2817-2828. |
| [12] | 姚远, 邓利君, 胡娟, 唐晓雨, 王铁, 李航, 孙国超, 熊博, 廖玲, 汪志辉. ‘脆红李’及其早熟芽变全基因组重测序分析[J]. 园艺学报, 2024, 51(10): 2255-2266. |
| [13] | 李春牛, 苏群, 李先民, 黄展文, 孙明艳, 卢家仕, 王虹妍, 卜朝阳. 茉莉花全基因组SSR标记开发及其亲缘关系鉴定[J]. 园艺学报, 2024, 51(10): 2343-2357. |
| [14] | 张力岚, 杨军, 王让剑. 茶树苯乙醇樱草糖苷含量相关遗传位点的挖掘[J]. 园艺学报, 2024, 51(1): 77-90. |
| [15] | 聂兴华, 张煜, 刘松, 杨佳宾, 郝雅琼, 刘阳, 秦岭, 邢宇. 基于基因组重测序的野生板栗遗传特征和分类地位研究[J]. 园艺学报, 2023, 50(8): 1622-1636. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2012 《园艺学报》编辑部 京ICP备10030308号-2 国际联网备案号 11010802023439
编辑部地址: 北京市海淀区中关村南大街12号中国农业科学院蔬菜花卉研究所 邮编: 100081
电话: 010-82109523 E-Mail: yuanyixuebao@126.com
技术支持:北京玛格泰克科技发展有限公司