
园艺学报 ›› 2024, Vol. 51 ›› Issue (1): 203-212.doi: 10.16420/j.issn.0513-353x.2023-0170
关夏玉1,2, 陈细红3, 郭菁3, 吕浩阳1, 梁晨媛1, 高芳銮1,*( )
)
收稿日期:2023-05-11
修回日期:2023-08-30
出版日期:2024-01-25
发布日期:2024-01-16
通讯作者:
基金资助:
GUAN Xiayu1,2, CHEN Xihong3, GUO Jing3, LÜ Haoyang1, LIANG Chenyuan1, GAO Fangluan1,*( )
)
Received:2023-05-11
Revised:2023-08-30
Published:2024-01-25
Online:2024-01-16
摘要:
建兰花叶病毒(Cymbidium mosaic virus,CymMV)是危害兰花并造成重要经济损失的病毒之一。为揭示CymMV的遗传变异及其适应性进化特征,新测定了16个CymMV石斛兰(Dendrobium nobile)分离物的外壳蛋白基因(coat protein,CP)序列,并结合已报道的52个CymMV中国分离物CP基因序列进行遗传变异及进化分析。遗传多样性分析结果显示,68个CymMV分离物CP基因的核苷酸多样性(Pi)和单倍型多样性(Hd)平均值分别为0.039和0.992,表明该病毒具有较高的遗传多样性。分子变异分析(analysis of molecular variance,AMOVA)显示,CymMV变异源主要来自于病毒个体,约占总变异的71.72%。群体遗传结构分析显示,该病毒可以分为两个大簇(Cluster),CymMV建兰(Cymbidium ensifolium)分离物独立为一簇(Cluster 1),其他寄主分离物聚为另一大簇(Cluster 2),Cluster 2内相同寄主来源的病毒分离物倾向于相聚成簇。进一步的系统发育与性状关联分析结果显示CymMV与寄主来源存在显著的关联性。上述结果表明,CymMV的遗传变异具有寄主特异性,其适应性进化主要受寄主因素所驱动。
关夏玉, 陈细红, 郭菁, 吕浩阳, 梁晨媛, 高芳銮. 建兰花叶病毒的遗传变异及其寄主适应性[J]. 园艺学报, 2024, 51(1): 203-212.
GUAN Xiayu, CHEN Xihong, GUO Jing, LÜ Haoyang, LIANG Chenyuan, GAO Fangluan. Genetic Variability and Host Adaption of Cymbidium Mosaic Virus[J]. Acta Horticulturae Sinica, 2024, 51(1): 203-212.
| 寄主 Host | 采样省份 Location | 分离物 Isolate | GenBank 登录号 Accession No. | 寄主 Host | 采样省份 Location | 分离物 Isolate | GenBank 登录号 Accession No. | |
|---|---|---|---|---|---|---|---|---|
| 国兰 Cymbidium sinense | 福建Fujian | CHN-FJZZ12 | DQ067878 | 蝴蝶兰 Phalaenopsis aphrodite | 河南Henan | CHN-HNZZ02 | KU873001 | |
| CHN-FJZZ13 | DQ067879 | 蕙兰C. faberi | 西藏Tibet | C-LZ02 | KP137368 | |||
| 湖北Hubei | CHN-HBWH21 | DQ067880 | C-LZ06 | KP137369 | ||||
| CHN-HBWH22 | DQ067881 | C-LZ14 | KP137370 | |||||
| 浙江Zhejiang | CHN-ZJHZ52 | DQ067882 | C-LZ18 | KP137371 | ||||
| CHN-ZJHZ61 | DQ067883 | C-LZ21 | KP137372 | |||||
| CHN-ZJHZ62 | DQ067884 | 石斛兰 Dendrobium nobile | 中国台湾 Taiwan,China | CyMV-1 * | OQ615774 | |||
| CHN-ZJHZ63 | DQ067885 | CyMV-2 * | OQ615782 | |||||
| CHN-ZJHZ67 | DQ067886 | CyMV-3 * | OQ615783 | |||||
| CHN-ZJHZ69 | DQ067887 | CyMV-4 * | OQ615784 | |||||
| CHN-ZJHZ612 | DQ067888 | CyMV-5 * | OQ615785 | |||||
| 蝴蝶兰 Phalaenopsis aphrodite | 浙江Zhejiang | CyMV-hangzhou | AM398153 | CyMV-6 * | OQ615786 | |||
| CrMV-HZ | DQ915439 | CyMV-7 * | OQ615787 | |||||
| 福建Fujian | xm | GQ507023 | CyMV-8 * | OQ615788 | ||||
| 江苏Jiangsu | CrMV-NJ-1 | JQ860108 | CyMV-9 * | OQ615789 | ||||
| 广东Guangdong | FS2 | KF853247 | CyMV-10 * | OQ615775 | ||||
| FS3 | KF853248 | CyMV-11 * | OQ615776 | |||||
| FS4 | KF853249 | CyMV-12 * | OQ615777 | |||||
| FS5 | KF853250 | CyMV-13* | OQ615778 | |||||
| GZ1 | KF853251 | CyMV-14 * | OQ615779 | |||||
| CH | KF853252 | CyMV-15 * | OQ615780 | |||||
| GZ2 | KF853253 | CyMV-16 * | OQ615781 | |||||
| GZ3 | KF853254 | 广东Guangdong | ZHD1 | EU672821 | ||||
| FS6 | KF853255 | 建兰 C. ensifolium | 浙江Zhejiang | ZJXTHC1 | GU295167 | |||
| ZH1 | KF853256 | 四川Sichuan | SCJJC7 | GU295168 | ||||
| ZH2 | KF853257 | SCDXZC4 | GU295172 | |||||
| FS7 | KF853258 | 广东 Guangdong | GZKTLC6 | GU295170 | ||||
| FS8 | KF853259 | GDDHLC5 | GU295173 | |||||
| FS9 | KF853260 | 北京 Beijing | BJKTLC3 | GU295169 | ||||
| FS10 | KF853261 | 云南 Yunnan | KMMLC2 | GU295171 | ||||
| SZ1 | KF853262 | 墨兰 C. haematodes | 广东 Guangdong | gd2 | AY360409 | |||
| SZ2 | KF853263 | gd3 | AY360410 | |||||
| SZ3 | KF853264 | 中国台湾 Taiwan,China | CrMV-CS | AY429021 | ||||
| SZ4 | KF853265 | Taiwan,China | AY571289 | |||||
表1 本研究中使用到的CymMV分离物
Table 1 CymMV isolates used in this study
| 寄主 Host | 采样省份 Location | 分离物 Isolate | GenBank 登录号 Accession No. | 寄主 Host | 采样省份 Location | 分离物 Isolate | GenBank 登录号 Accession No. | |
|---|---|---|---|---|---|---|---|---|
| 国兰 Cymbidium sinense | 福建Fujian | CHN-FJZZ12 | DQ067878 | 蝴蝶兰 Phalaenopsis aphrodite | 河南Henan | CHN-HNZZ02 | KU873001 | |
| CHN-FJZZ13 | DQ067879 | 蕙兰C. faberi | 西藏Tibet | C-LZ02 | KP137368 | |||
| 湖北Hubei | CHN-HBWH21 | DQ067880 | C-LZ06 | KP137369 | ||||
| CHN-HBWH22 | DQ067881 | C-LZ14 | KP137370 | |||||
| 浙江Zhejiang | CHN-ZJHZ52 | DQ067882 | C-LZ18 | KP137371 | ||||
| CHN-ZJHZ61 | DQ067883 | C-LZ21 | KP137372 | |||||
| CHN-ZJHZ62 | DQ067884 | 石斛兰 Dendrobium nobile | 中国台湾 Taiwan,China | CyMV-1 * | OQ615774 | |||
| CHN-ZJHZ63 | DQ067885 | CyMV-2 * | OQ615782 | |||||
| CHN-ZJHZ67 | DQ067886 | CyMV-3 * | OQ615783 | |||||
| CHN-ZJHZ69 | DQ067887 | CyMV-4 * | OQ615784 | |||||
| CHN-ZJHZ612 | DQ067888 | CyMV-5 * | OQ615785 | |||||
| 蝴蝶兰 Phalaenopsis aphrodite | 浙江Zhejiang | CyMV-hangzhou | AM398153 | CyMV-6 * | OQ615786 | |||
| CrMV-HZ | DQ915439 | CyMV-7 * | OQ615787 | |||||
| 福建Fujian | xm | GQ507023 | CyMV-8 * | OQ615788 | ||||
| 江苏Jiangsu | CrMV-NJ-1 | JQ860108 | CyMV-9 * | OQ615789 | ||||
| 广东Guangdong | FS2 | KF853247 | CyMV-10 * | OQ615775 | ||||
| FS3 | KF853248 | CyMV-11 * | OQ615776 | |||||
| FS4 | KF853249 | CyMV-12 * | OQ615777 | |||||
| FS5 | KF853250 | CyMV-13* | OQ615778 | |||||
| GZ1 | KF853251 | CyMV-14 * | OQ615779 | |||||
| CH | KF853252 | CyMV-15 * | OQ615780 | |||||
| GZ2 | KF853253 | CyMV-16 * | OQ615781 | |||||
| GZ3 | KF853254 | 广东Guangdong | ZHD1 | EU672821 | ||||
| FS6 | KF853255 | 建兰 C. ensifolium | 浙江Zhejiang | ZJXTHC1 | GU295167 | |||
| ZH1 | KF853256 | 四川Sichuan | SCJJC7 | GU295168 | ||||
| ZH2 | KF853257 | SCDXZC4 | GU295172 | |||||
| FS7 | KF853258 | 广东 Guangdong | GZKTLC6 | GU295170 | ||||
| FS8 | KF853259 | GDDHLC5 | GU295173 | |||||
| FS9 | KF853260 | 北京 Beijing | BJKTLC3 | GU295169 | ||||
| FS10 | KF853261 | 云南 Yunnan | KMMLC2 | GU295171 | ||||
| SZ1 | KF853262 | 墨兰 C. haematodes | 广东 Guangdong | gd2 | AY360409 | |||
| SZ2 | KF853263 | gd3 | AY360410 | |||||
| SZ3 | KF853264 | 中国台湾 Taiwan,China | CrMV-CS | AY429021 | ||||
| SZ4 | KF853265 | Taiwan,China | AY571289 | |||||
图2 CymMV CP基因的RT-PCR扩增结果 10 ~ 12:阳性对照、阴性对照和空白对照。
Fig. 2 RT-PCR from the amplification of CP gene of CymMV 10–12:Positive control,negative control and blank control.
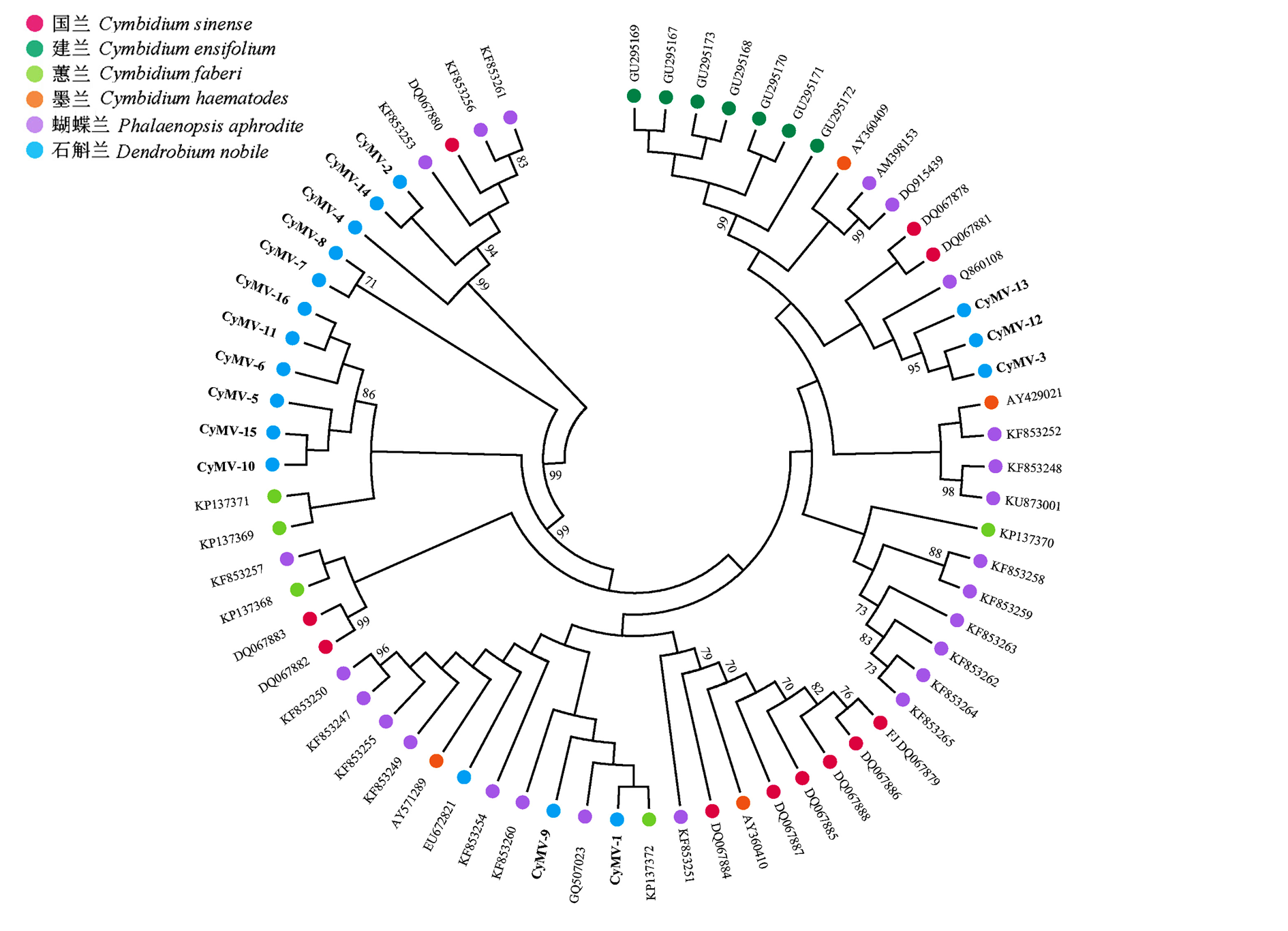
图3 基于CymMV CP基因核苷酸序列重建的系统发育树 分支节点上的数值为自举值(仅显示 > 50%),不同颜色指示的圆点为不同寄主来源的分离物。
Fig. 3 Phylogenetic tree reconstructed from the CP gene sequence of CymMV Numbers above branches indicate the bootstrap values(only shown > 50%),and CymMV isolates from different host species are indicated by circles with differences in colours.
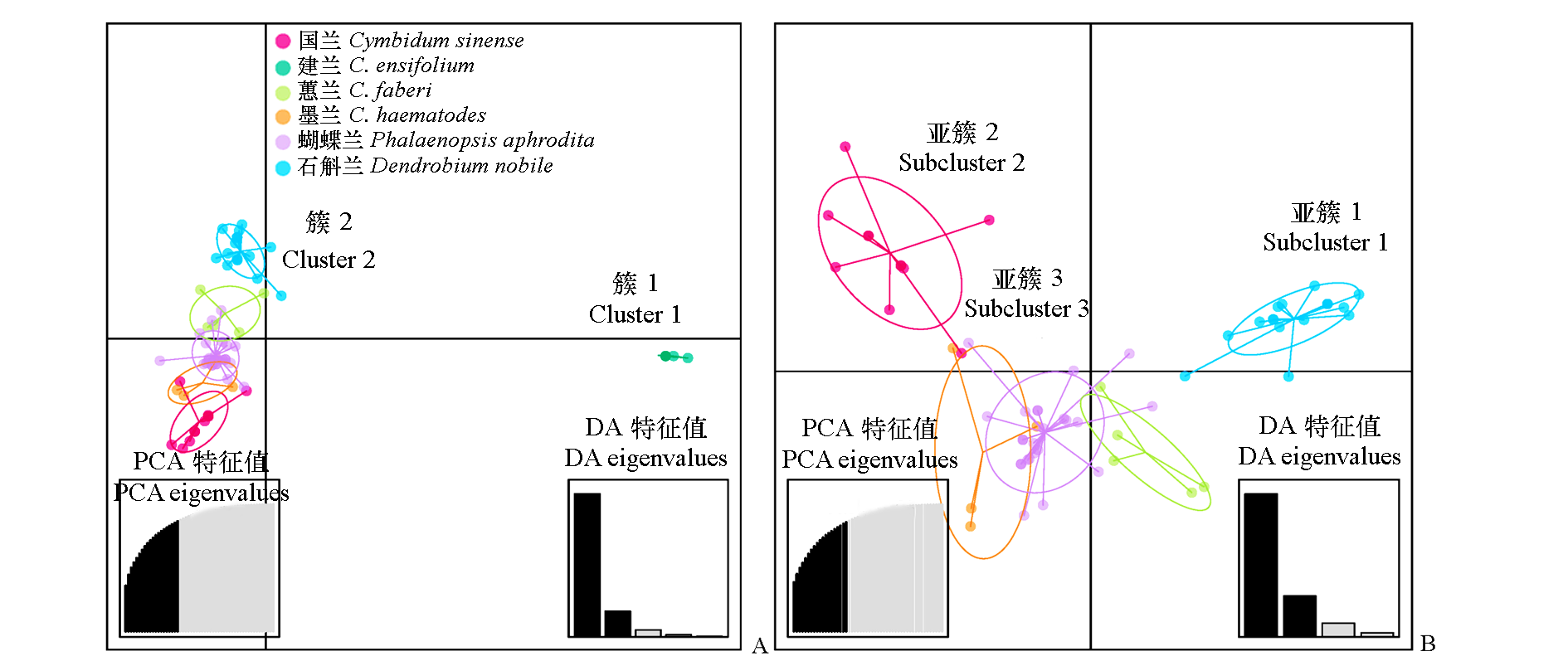
图4 主成分判别分析(DAPC)结果 A:68 个CymMV分离物的聚类散点图;B:Cluster 2中CymMV分离物的聚类散点图。
Fig. 4 Results from the discriminant analysis of principal components(DAPC) A:Scatterplots for 68 CymMV isolates; B:Scatterplots for CymMV isolates within Cluster 2.
| 簇 Cluster | 亚簇 Subcluster | 样本量大小 Sample size | 单倍型 Haplotypes | 单倍型多样性Hd Haplotype diversity | 核苷酸多样性Pi Nucleotide diversity |
|---|---|---|---|---|---|
| 1 | 7 | 3 | 0.524 ± 0.209 | 0.002 ± 0.009 | |
| 2 | 61 | 53 | 0.995 ± 0.004 | 0.040 ± 0.004 | |
| 2 | 1 | 17 | 14 | 0.971 ± 0.032 | 0.041 ± 0.008 |
| 2 | 11 | 9 | 0.961 ± 0.051 | 0.041 ± 0.012 | |
| 3 | 33 | 30 | 0.994 ± 0.009 | 0.036 ± 0.007 | |
| 总计Total | 68 | 56 | 0.992 ± 0.005 | 0.039 ± 0.004 |
表2 CymMV CP基因的遗传多样性
Table 2 Summary statistics for genetic diversity of the CP gene of CymMV
| 簇 Cluster | 亚簇 Subcluster | 样本量大小 Sample size | 单倍型 Haplotypes | 单倍型多样性Hd Haplotype diversity | 核苷酸多样性Pi Nucleotide diversity |
|---|---|---|---|---|---|
| 1 | 7 | 3 | 0.524 ± 0.209 | 0.002 ± 0.009 | |
| 2 | 61 | 53 | 0.995 ± 0.004 | 0.040 ± 0.004 | |
| 2 | 1 | 17 | 14 | 0.971 ± 0.032 | 0.041 ± 0.008 |
| 2 | 11 | 9 | 0.961 ± 0.051 | 0.041 ± 0.012 | |
| 3 | 33 | 30 | 0.994 ± 0.009 | 0.036 ± 0.007 | |
| 总计Total | 68 | 56 | 0.992 ± 0.005 | 0.039 ± 0.004 |
| 变异源 Source of variation | 自由度 df | 平方和 Sum of squares | 变量组分 Variance components | 变异比例 Percentage of variation | 固定系数 Fixation index |
|---|---|---|---|---|---|
| 簇间Among clusters | 1 | 65.903 | 3.580 | 21.880 | 0.220 ns |
| 簇内亚簇间Among sub-clusters within clusters | 2 | 61.563 | 1.046 | 6.390 | 0.082* |
| 亚簇内Within sub-clusters | 64 | 750.931 | 11.733 | 71.720 | 0.282*** |
| 总计Total | 67 | 878.397 | 16.359 |
表3 CymMV CP基因的分子变异分析
Table 3 Hierarchical analysis of molecular variance for the CP gene of CymMV
| 变异源 Source of variation | 自由度 df | 平方和 Sum of squares | 变量组分 Variance components | 变异比例 Percentage of variation | 固定系数 Fixation index |
|---|---|---|---|---|---|
| 簇间Among clusters | 1 | 65.903 | 3.580 | 21.880 | 0.220 ns |
| 簇内亚簇间Among sub-clusters within clusters | 2 | 61.563 | 1.046 | 6.390 | 0.082* |
| 亚簇内Within sub-clusters | 64 | 750.931 | 11.733 | 71.720 | 0.282*** |
| 总计Total | 67 | 878.397 | 16.359 |
| 簇 Cluster | 亚簇 Subcluster | Tajima's D | P值 P value | Fu's FS | P 值 P value |
|---|---|---|---|---|---|
| 1 | -1.434 | 0.067 | 0.263 | 0.475ns | |
| 2 | -1.335 | 0.043* | -19.765 | 0.000*** | |
| 2 | 1 | 0.483 | 0.729 | 0.029 | 0.517 ns |
| 2 | -1.446 | 0.072 | 1.339 | 0.696 ns | |
| 3 | -1.309 | 0.042* | -8.222 | 0.011* |
表4 不同CymMV群体的中性检验
Table 4 Neutrality tests for different CymMV populations
| 簇 Cluster | 亚簇 Subcluster | Tajima's D | P值 P value | Fu's FS | P 值 P value |
|---|---|---|---|---|---|
| 1 | -1.434 | 0.067 | 0.263 | 0.475ns | |
| 2 | -1.335 | 0.043* | -19.765 | 0.000*** | |
| 2 | 1 | 0.483 | 0.729 | 0.029 | 0.517 ns |
| 2 | -1.446 | 0.072 | 1.339 | 0.696 ns | |
| 3 | -1.309 | 0.042* | -8.222 | 0.011* |
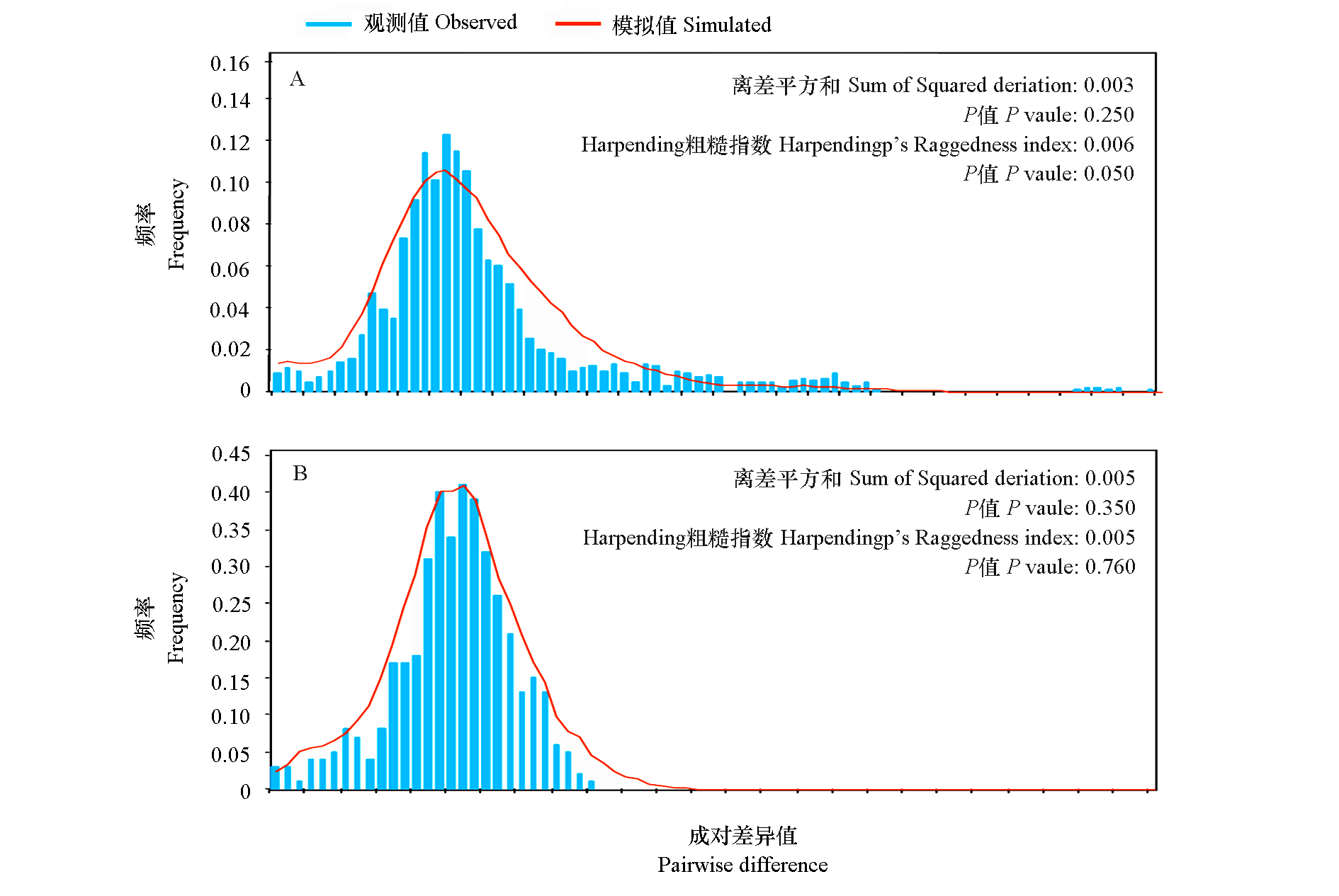
图5 不同簇或亚簇的错配分布分析结果 A:CymMV簇2;B:CymMV簇2内的亚簇3。
Fig. 5 Mismatch distribution analyses for different clusters or subcluster 3 of CymMV A:Cluster 2 of CymMV;B:Subcluster 3 within Cluster 2 of CymMV.
| 统计参数Statistic | 寄主 Host | 观测平均值(95% HPD) Observed mean(95% HPD) | 零假设平均值(95% HPD) Null mean(95% HPD) | P 值 P value |
|---|---|---|---|---|
| AI | 1.319(0.830 ~ 1.803) | 5.775(5.005 ~ 6.531) | < 0.001*** | |
| PS | 20.472(19.000 ~ 21.000) | 38.091(35.370 ~ 0.624) | < 0.001*** | |
| MC | 国兰Cymbidium sinense | 4.064(3.000 ~ 5.000) | 1.518(1.003 ~ 2.182) | 0.010** |
| 建兰C. ensifolium | 6.981(6.000 ~ 7.000) | 1.225(1.000 ~ 1.998) | 0.010** | |
| 蕙兰C. faberi | 2.049(2.000 ~ 3.000) | 1.103(1.000 ~ 1.989) | 0.030* | |
| 墨兰C. haematodes | 1.008(1.000 ~ 2.000) | 1.065(1.000 ~ 1.335) | 1.000ns | |
| 蝴蝶兰Phalaenopsis | 6.017(6.000 ~ 7.000) | 2.414(1.686 ~ 3.375) | 0.010** | |
| 石斛兰Dendrobium nobile | 5.975(5.000 ~ 6.000) | 1.887(1.079 ~ 2.982) | 0.010** |
表 5 CymMV遗传多样性的寄主BaTS关联分析
Table 5 Bayesian Tip-association significance(BaTS)testing for the host effect on the genetic diversity of CymMV isolates
| 统计参数Statistic | 寄主 Host | 观测平均值(95% HPD) Observed mean(95% HPD) | 零假设平均值(95% HPD) Null mean(95% HPD) | P 值 P value |
|---|---|---|---|---|
| AI | 1.319(0.830 ~ 1.803) | 5.775(5.005 ~ 6.531) | < 0.001*** | |
| PS | 20.472(19.000 ~ 21.000) | 38.091(35.370 ~ 0.624) | < 0.001*** | |
| MC | 国兰Cymbidium sinense | 4.064(3.000 ~ 5.000) | 1.518(1.003 ~ 2.182) | 0.010** |
| 建兰C. ensifolium | 6.981(6.000 ~ 7.000) | 1.225(1.000 ~ 1.998) | 0.010** | |
| 蕙兰C. faberi | 2.049(2.000 ~ 3.000) | 1.103(1.000 ~ 1.989) | 0.030* | |
| 墨兰C. haematodes | 1.008(1.000 ~ 2.000) | 1.065(1.000 ~ 1.335) | 1.000ns | |
| 蝴蝶兰Phalaenopsis | 6.017(6.000 ~ 7.000) | 2.414(1.686 ~ 3.375) | 0.010** | |
| 石斛兰Dendrobium nobile | 5.975(5.000 ~ 6.000) | 1.887(1.079 ~ 2.982) | 0.010** |
| [1] |
|
|
常飞, 邹文超, 高芳銮, 沈建国, 詹家绥. 2015. 不同寄主来源的马铃薯Y病毒群体遗传结构的比较分析. 遗传, 37 (3): 292-301.
|
|
| [2] |
doi: 10.16420/j.issn.0513-353x.2020-1053 URL |
|
陈健鑫, 魏玉倩, 唐婕, 竺永金, 马焕成, 伍建榕. 2022. 云南大理栽培春兰中ORSV和CymMV的分子检测及序列分析. 园艺学报, 49 (3):655-662.
doi: 10.16420/j.issn.0513-353x.2020-1053 URL |
|
| [3] |
doi: 10.1099/vir.0.044347-0 pmid: 22837421 |
| [4] |
doi: 10.1111/j.1755-0998.2010.02847.x pmid: 21565059 |
| [5] |
|
| [6] |
doi: 10.1111/eva.2017.10.issue-4 URL |
| [7] |
doi: 10.3389/fmicb.2022.1051834 URL |
| [8] |
|
| [9] |
doi: 10.1016/j.ympev.2021.107336 URL |
| [10] |
doi: 10.1007/s11262-011-0592-x URL |
| [11] |
doi: 10.1186/1471-2156-11-94 URL |
| [12] |
doi: 10.1099/jgv.0.001436 pmid: 32525472 |
| [13] |
doi: 10.1093/molbev/msy096 URL |
| [14] |
doi: 10.16420/j.issn.0513-353x.2021-0886 URL |
|
李世帆, 邵宇纯, 祁豪男, 杨兰, 李小伟, 徐秉良, 陈雅寒. 2022. 甘肃省苹果坏死花叶病毒检测及遗传多样性分析. 园艺学报, 49 (11):2431-2438.
doi: 10.16420/j.issn.0513-353x.2021-0886 URL |
|
| [15] |
doi: 10.1093/bioinformatics/btp187 pmid: 19346325 |
| [16] |
doi: 10.1007/s00705-016-2773-3 pmid: 26923929 |
| [17] |
doi: 10.1093/ve/vev003 URL |
| [18] |
doi: 10.1007/s13205-019-1876-4 |
| [19] |
|
|
明艳林, 李梅, 郑国华. 2005. 建兰花叶病毒研究进展. 福建农业学报, 20 (1):30-33.
|
|
| [20] |
doi: 10.1007/s00705-006-0897-6 pmid: 17216134 |
| [21] |
doi: 10.1111/mpp.v21.2 URL |
| [22] |
doi: 10.1016/j.meegid.2007.08.001 URL |
| [23] |
doi: 10.1093/sysbio/sys029 pmid: 22357727 |
| [24] |
doi: 10.1093/molbev/msv222 pmid: 26503941 |
| [25] |
|
|
魏永路, 任锐, 高洁, 刘伟平, 谢琦, 朱根发, 杨凤玺. 2022. 建兰花叶病毒广东分离物的全基因组序列分析. 广东农业科学, 49 (2):109-117.
|
|
| [26] |
|
|
肖火根, 郑冠标, 张曙光, 高乔婉. 1996. 广东兰花病毒病的鉴定和检测研究. 华南农业大学学报, 17 (1):21-24.
|
|
| [27] |
|
|
谢联辉, 林奇英. 2011. 植物病毒学(第3版). 北京: 中国农业出版社.
|
|
| [28] |
doi: 10.1007/s11262-010-0468-5 URL |
| [29] |
|
| [30] |
doi: 10.3389/fmicb.2021.738646 URL |
| [1] | 毕清芮, 崔东升, 马新院, 薛玉冉, 章世奎, 樊国全, 牛莹莹. 新疆本地梨品种的遗传多样性及亲缘关系[J]. 园艺学报, 2025, 52(3): 561-574. |
| [2] | 罗嗣芳, 严翔, 谢丽芳, 孙静贤, 郭紫晶, 张祖铭, 陈兆星. 赣南野生山金柑全基因组InDel标记开发及应用[J]. 园艺学报, 2025, 52(2): 267-278. |
| [3] | 张露, 张平萍, 解玮佳, 许凤, 彭绿春, 宋杰, 杜光辉, 李世峰. 常绿杜鹃种质遗传多样性分析及SSR指纹图谱构建[J]. 园艺学报, 2025, 52(2): 380-394. |
| [4] | 肖熙鸥, 聂珩, 林文秋, 吴彩玉. 茄子全基因组SNP标记的开发[J]. 园艺学报, 2025, 52(1): 88-100. |
| [5] | 秦子璐, 徐正康, 戴晓港, 陈赢男. 望春玉兰种质资源遗传多样性分析与核心种质构建[J]. 园艺学报, 2024, 51(8): 1823-1832. |
| [6] | 袁娜, 徐勤圆, 徐照龙, 周玲, 刘晓庆, 陈新, 杜建厂. 基于靶向测序技术的菜豆SNP液相芯片开发及验证[J]. 园艺学报, 2024, 51(5): 1017-1032. |
| [7] | 游平, 杨进, 周俊, 黄爱军, 鲍敏丽, 易龙. 柑橘黄龙病菌原噬菌体的遗传多样性分析[J]. 园艺学报, 2024, 51(4): 727-736. |
| [8] | 熊志伟, 李智龙, 尹晖, 高玉霞. 柑橘黄龙病菌亚洲种的泛基因组分析[J]. 园艺学报, 2024, 51(4): 737-747. |
| [9] | 尤园园, 王帅, 方莹莹, 齐诚, 李淑培, 张映, 陈钰辉, 刘伟, 刘富中, 舒金帅. 茄子全基因组InDel变异特征及分子标记开发和应用[J]. 园艺学报, 2024, 51(3): 520-532. |
| [10] | 陈方策, 胡平正, 解为玮, 王钲彭, 钟创南, 李海炎, 何业华, 彭泽, 万保雄, 刘朝阳. 基于重测序的中国李基因组InDel标记的开发及应用[J]. 园艺学报, 2024, 51(11): 2510-2522. |
| [11] | 王荣波, 王海云, 张前荣, 蔡松龄, 李本金, 张美祥, 刘裴清. 福建省番茄青枯病菌遗传多样性分析及其抗性砧木的筛选[J]. 园艺学报, 2024, 51(11): 2540-2554. |
| [12] | 李春牛, 苏群, 李先民, 黄展文, 孙明艳, 卢家仕, 王虹妍, 卜朝阳. 茉莉花全基因组SSR标记开发及其亲缘关系鉴定[J]. 园艺学报, 2024, 51(10): 2343-2357. |
| [13] | 裴芸, 虞夏清, 赵晓坤, 张万萍, 陈劲枫. 多倍化与植物新表型关联性的研究进展[J]. 园艺学报, 2023, 50(9): 1854-1866. |
| [14] | 王瑞, 洪文娟, 罗华, 赵丽娜, 陈颖, 王君. 石榴品种SSR指纹图谱构建及杂种父本鉴定[J]. 园艺学报, 2023, 50(2): 265-278. |
| [15] | 王梦梦, 孙德岭, 陈锐, 杨迎霞, 张冠, 吕明杰, 王倩, 谢添羽, 牛国保, 单晓政, 谭津, 姚星伟. 花椰菜核心种质的构建与评价[J]. 园艺学报, 2023, 50(2): 421-431. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2012 《园艺学报》编辑部 京ICP备10030308号-2 国际联网备案号 11010802023439
编辑部地址: 北京市海淀区中关村南大街12号中国农业科学院蔬菜花卉研究所 邮编: 100081
电话: 010-82109523 E-Mail: yuanyixuebao@126.com
技术支持:北京玛格泰克科技发展有限公司