
园艺学报 ›› 2024, Vol. 51 ›› Issue (3): 545-559.doi: 10.16420/j.issn.0513-353x.2023-0098
刘晋红, 王峥, 于昊, 辛依睿, 亓果宁, 柳参奎, 任慧敏*( )
)
收稿日期:2023-04-16
修回日期:2023-11-17
出版日期:2024-03-25
发布日期:2024-03-22
通讯作者:
基金资助:
LIU Jinhong, WANG Zheng, YU Hao, XIN Yirui, QI Guoning, LIU Shenkui, REN Huimin*( )
)
Received:2023-04-16
Revised:2023-11-17
Published:2024-03-25
Online:2024-03-22
摘要:
为了研究S型阴离子通道蛋白(slow type anion channel,SLAC)基因家族在毛竹(Phyllostachys edulis)响应干旱胁迫中的作用,利用生物信息方法以拟南芥和水稻中的SLAC/SLAH家族蛋白序列为模板对毛竹中SLAC家族成员进行鉴定,对其基因结构、进化关系和组织表达等进行分析,并对PheSLAC1的保守功能位点以及在干旱胁迫中的功能进行初步研究。结果表明:毛竹中包含15个SLAC家族基因,其中PheSLAC1.1(PH02Gene01933.t1)和PheSLAC1.2(PH02Gene00592.t1)在叶片中表达量最高,基因编码区(coding sequence,CDS)序列长度分别为1 716和1 677 bp,分别编码572和559个氨基酸,且包含保守的关键功能位点。亚细胞定位结果表明,PheSLAC1.1和PheSLAC1.2均定位于细胞质膜中。PheSLAC1.1和PheSLAC1.2在拟南芥slac1-3突变体中的表达结果表明,其能够部分减轻突变体的干旱敏感表型,表明两者均能够通过调控气孔关闭在毛竹响应干旱胁迫中发挥重要作用。
刘晋红, 王峥, 于昊, 辛依睿, 亓果宁, 柳参奎, 任慧敏. 毛竹SLAC家族基因鉴定及PheSLAC1功能分析[J]. 园艺学报, 2024, 51(3): 545-559.
LIU Jinhong, WANG Zheng, YU Hao, XIN Yirui, QI Guoning, LIU Shenkui, REN Huimin. Identification of SLAC Gene Family in Phyllostachys edulis and Characterization of PheSLAC1[J]. Acta Horticulturae Sinica, 2024, 51(3): 545-559.
| 引物用途 Primer use | 引物名称 Primer name | 引物序列(5′-3′) Primer sequence | |
|---|---|---|---|
| 实时荧光定量引物 qRT-PCR primer | NTB | F:TCTTGTTTGACACCGAAGAGGAG; | R:AATAGCTGTCCCTGGAGGAGTTT |
| PheSLAC1.1 | F:FTTCTTCATGGCGCTCTTCTT; | R:GACATGAGGGAGAGGCTCAG | |
| PheSLAC1.2 | F:GCGCGGACATTATTCTTCAT; | R:GACATGAGGGAGAGGCTCAG | |
| PH02Gene35134 | F:ATCTACGGCCAGTGGTTCAC; | R:TGACGAACAGCACGAGGTAG | |
| PH02Gene08796 | F:CATGAACTACCTCTTCGCGC; | R:GAACCACTGGCCGTAGATCT | |
| PH02Gene45054 | F:ACTACAGCGTGCTGTTCGTG; | R:GAACCCACGGAAGAAGTTGA | |
| PH02Gene22907 | F:TGTTCGTGACGCTCTACCAG; | R:TGAACCCCCTGAAAAAGTTG | |
| PH02Gene22131 | F:CCAGAGTCCAACGCTTCTTC; | R:ATCTTGAGCGTGATGTGCAG | |
| PH02Gene10869 | F:AGCTAAGGTCGGAGGAGGAG; | R:ATCTTGAGCGTGATGTGCAG | |
| PH02Gene20948 | F:ATCTACGGGCAGTGGATGTC; | R:TGGTACAGCGTGACGAAGAG | |
| PH02Gene48421 | F:GCCTCCCTGAATGAGCATAA; | R:ATGCTGAGCGAAAGGGAGTA | |
| PH02Gene25481 | F:GTGGATGAACAGGGGAAAGA; | R:GGACCTCGAGTTTTGTCTCG | |
| PH02Gene41494 | F:GGAAGGACGTCGTGATTAGC; | R:GGACCTCGAGTTTTGTCTCG | |
| PH02Gene06334 | F:CTGTCCGCCGAGAGTTTTAC; | R:GAGCTCAAGGCAGAAGATGG | |
| PH02Gene06335 | F:GGCTTCGTCTCGGTGATCTA; | R:CATCCACTGCCCGTAGATCT | |
| PH02Gene20553 | F:ATCTACGGGCAGTGGATGTC; | R:TGACGAACAGCACGAGGTAG | |
| AtACTIN2 | F:GGTAACATTGTGCTCAGTGGTGG; | R:AACGACCTTAATCTTCATGCTGC | |
| 转基因和亚细胞定位 Transgene and subcellular localization | PheSLAC1.1-1304 | F: GAAGATCTTATGGCAGCCGAGCCATCGTC R: CGGACTAGTGTCTGTTTTCTCCTCTTCGTCCTT | |
| PheSLAC1.2-1304 | F: GAAGATCTTATGGCAGCCGAGCCATCGTC R: CGGACTAGTTTCTGTTTCCTCCTCTTCATCCTTG | ||
| PheSLAC1.1-GFP | F: TCCCCCCGGGATGGCAGCCGAGCCATCGTCT R: CTAGTCTAGAGTCTGTTTTCTCCTCTTCGTCCTT | ||
| PheSLAC1.2-GFP | F: TCCCCCCGGGATGGCAGCCGAGCCATCGTC R: CTAGTCT AGATTCTGTTTCCTCCTCTTCATCCT | ||
表1 引物序列
Table 1 Primers used in this study
| 引物用途 Primer use | 引物名称 Primer name | 引物序列(5′-3′) Primer sequence | |
|---|---|---|---|
| 实时荧光定量引物 qRT-PCR primer | NTB | F:TCTTGTTTGACACCGAAGAGGAG; | R:AATAGCTGTCCCTGGAGGAGTTT |
| PheSLAC1.1 | F:FTTCTTCATGGCGCTCTTCTT; | R:GACATGAGGGAGAGGCTCAG | |
| PheSLAC1.2 | F:GCGCGGACATTATTCTTCAT; | R:GACATGAGGGAGAGGCTCAG | |
| PH02Gene35134 | F:ATCTACGGCCAGTGGTTCAC; | R:TGACGAACAGCACGAGGTAG | |
| PH02Gene08796 | F:CATGAACTACCTCTTCGCGC; | R:GAACCACTGGCCGTAGATCT | |
| PH02Gene45054 | F:ACTACAGCGTGCTGTTCGTG; | R:GAACCCACGGAAGAAGTTGA | |
| PH02Gene22907 | F:TGTTCGTGACGCTCTACCAG; | R:TGAACCCCCTGAAAAAGTTG | |
| PH02Gene22131 | F:CCAGAGTCCAACGCTTCTTC; | R:ATCTTGAGCGTGATGTGCAG | |
| PH02Gene10869 | F:AGCTAAGGTCGGAGGAGGAG; | R:ATCTTGAGCGTGATGTGCAG | |
| PH02Gene20948 | F:ATCTACGGGCAGTGGATGTC; | R:TGGTACAGCGTGACGAAGAG | |
| PH02Gene48421 | F:GCCTCCCTGAATGAGCATAA; | R:ATGCTGAGCGAAAGGGAGTA | |
| PH02Gene25481 | F:GTGGATGAACAGGGGAAAGA; | R:GGACCTCGAGTTTTGTCTCG | |
| PH02Gene41494 | F:GGAAGGACGTCGTGATTAGC; | R:GGACCTCGAGTTTTGTCTCG | |
| PH02Gene06334 | F:CTGTCCGCCGAGAGTTTTAC; | R:GAGCTCAAGGCAGAAGATGG | |
| PH02Gene06335 | F:GGCTTCGTCTCGGTGATCTA; | R:CATCCACTGCCCGTAGATCT | |
| PH02Gene20553 | F:ATCTACGGGCAGTGGATGTC; | R:TGACGAACAGCACGAGGTAG | |
| AtACTIN2 | F:GGTAACATTGTGCTCAGTGGTGG; | R:AACGACCTTAATCTTCATGCTGC | |
| 转基因和亚细胞定位 Transgene and subcellular localization | PheSLAC1.1-1304 | F: GAAGATCTTATGGCAGCCGAGCCATCGTC R: CGGACTAGTGTCTGTTTTCTCCTCTTCGTCCTT | |
| PheSLAC1.2-1304 | F: GAAGATCTTATGGCAGCCGAGCCATCGTC R: CGGACTAGTTTCTGTTTCCTCCTCTTCATCCTTG | ||
| PheSLAC1.1-GFP | F: TCCCCCCGGGATGGCAGCCGAGCCATCGTCT R: CTAGTCTAGAGTCTGTTTTCTCCTCTTCGTCCTT | ||
| PheSLAC1.2-GFP | F: TCCCCCCGGGATGGCAGCCGAGCCATCGTC R: CTAGTCT AGATTCTGTTTCCTCCTCTTCATCCT | ||
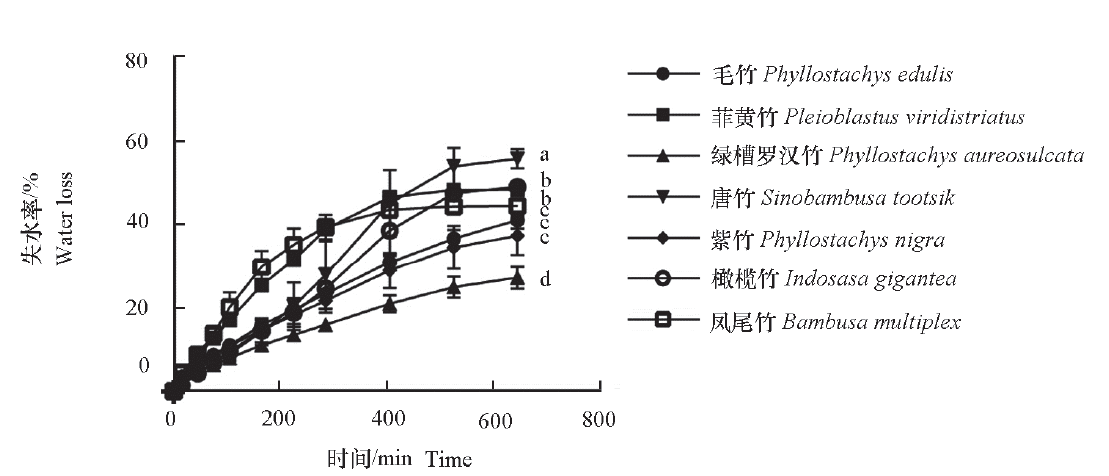
图1 不同竹离体叶片失水率变化 不同字母表示在 P < 0.05 水平差异显著。下同。
Fig. 1 Water loss rate of different bamboo species Different lowercase letters indicate significant differences at P < 0.05. The same below.
| 种类 Species | 蒸腾速率/(mmol · m-2 · s-1) Transpiration rate | 气孔导度/(mmol · m-2 · s-1) Stomatal conductance |
|---|---|---|
| 毛竹 Phyllostachys edulis | 0.938 | 0.058 |
| 菲黄竹 Pleioblastus viridistriatus | 1.198 | 0.073 |
| 绿槽罗汉竹 Phyllostachys aureosulcata | 0.592 | 0.037 |
| 唐竹 Sinobambusa tootsik | 0.482 | 0.029 |
| 紫竹 Phyllostachys nigra | 0.641 | 0.040 |
| 橄榄竹 Indosasa gigantea | 0.655 | 0.048 |
| 凤尾竹 Bambusa multiplex | 1.562 | 0.101 |
表2 不同竹叶片蒸腾速率和气孔导度
Table 2 Transpiration rate and stomatal conductance of different bamboo species
| 种类 Species | 蒸腾速率/(mmol · m-2 · s-1) Transpiration rate | 气孔导度/(mmol · m-2 · s-1) Stomatal conductance |
|---|---|---|
| 毛竹 Phyllostachys edulis | 0.938 | 0.058 |
| 菲黄竹 Pleioblastus viridistriatus | 1.198 | 0.073 |
| 绿槽罗汉竹 Phyllostachys aureosulcata | 0.592 | 0.037 |
| 唐竹 Sinobambusa tootsik | 0.482 | 0.029 |
| 紫竹 Phyllostachys nigra | 0.641 | 0.040 |
| 橄榄竹 Indosasa gigantea | 0.655 | 0.048 |
| 凤尾竹 Bambusa multiplex | 1.562 | 0.101 |
| 基因登录号 ID | 染色体位置 Chromosome location | 等电点 pI | 相对分子量/kD MW | 长度/aa Length | 跨膜区数量 No. of transmembrane domain |
|---|---|---|---|---|---|
| PH02Gene00592.t1 | hic_scaffold_24 | 9.63 | 61 635.52 | 558 | 9 |
| PH02Gene01933.t1 | hic_scaffold_23 | 9.94 | 62 657.88 | 571 | 10 |
| PH02Gene35134.t1 | hic_scaffold_16 | 10.18 | 33 127.26 | 294 | 9 |
| PH02Gene08796.t1 | hic_scaffold_6 | 9.87 | 42 370.12 | 384 | 10 |
| PH02Gene45054.t2 | hic_scaffold_9 | 9.13 | 64 409.35 | 586 | 10 |
| PH02Gene22907.t1 | hic_scaffold_18 | 8.52 | 67 869.30 | 621 | 10 |
| PH02Gene22131.t1 | hic_scaffold_16 | 9.26 | 64 301.36 | 583 | 10 |
| PH02Gene10869.t1 | hic_scaffold_14 | 9.18 | 67 749.68 | 609 | 10 |
| PH02Gene20948.t2 | hic_scaffold_16 | 8.82 | 72 183.69 | 651 | 10 |
| PH02Gene48421.t1 | hic_scaffold_14 | 8.70 | 68 667.45 | 613 | 10 |
| PH02Gene25481.t1 | hic_scaffold_342 | 8.02 | 73 583.66 | 663 | 9 |
| PH02Gene41494.t1 | hic_scaffold_9 | 7.71 | 69 005.90 | 623 | 9 |
| PH02Gene06334.t1 | hic_scaffold_7 | 7.03 | 70 670.36 | 640 | 8 |
| PH02Gene06335.t1 | hic_scaffold_7 | 7.30 | 70 425.26 | 640 | 10 |
| PH02Gene20553.t1 | hic_scaffold_13 | 7.09 | 77 827.50 | 708 | 9 |
表3 毛竹的SLAC1基因家族
Table 3 The SLAC1 gene family of moso bamboo
| 基因登录号 ID | 染色体位置 Chromosome location | 等电点 pI | 相对分子量/kD MW | 长度/aa Length | 跨膜区数量 No. of transmembrane domain |
|---|---|---|---|---|---|
| PH02Gene00592.t1 | hic_scaffold_24 | 9.63 | 61 635.52 | 558 | 9 |
| PH02Gene01933.t1 | hic_scaffold_23 | 9.94 | 62 657.88 | 571 | 10 |
| PH02Gene35134.t1 | hic_scaffold_16 | 10.18 | 33 127.26 | 294 | 9 |
| PH02Gene08796.t1 | hic_scaffold_6 | 9.87 | 42 370.12 | 384 | 10 |
| PH02Gene45054.t2 | hic_scaffold_9 | 9.13 | 64 409.35 | 586 | 10 |
| PH02Gene22907.t1 | hic_scaffold_18 | 8.52 | 67 869.30 | 621 | 10 |
| PH02Gene22131.t1 | hic_scaffold_16 | 9.26 | 64 301.36 | 583 | 10 |
| PH02Gene10869.t1 | hic_scaffold_14 | 9.18 | 67 749.68 | 609 | 10 |
| PH02Gene20948.t2 | hic_scaffold_16 | 8.82 | 72 183.69 | 651 | 10 |
| PH02Gene48421.t1 | hic_scaffold_14 | 8.70 | 68 667.45 | 613 | 10 |
| PH02Gene25481.t1 | hic_scaffold_342 | 8.02 | 73 583.66 | 663 | 9 |
| PH02Gene41494.t1 | hic_scaffold_9 | 7.71 | 69 005.90 | 623 | 9 |
| PH02Gene06334.t1 | hic_scaffold_7 | 7.03 | 70 670.36 | 640 | 8 |
| PH02Gene06335.t1 | hic_scaffold_7 | 7.30 | 70 425.26 | 640 | 10 |
| PH02Gene20553.t1 | hic_scaffold_13 | 7.09 | 77 827.50 | 708 | 9 |

图3 PheSLAC基因家族系统发育树(A)、保守基序(B)和保守结构域(C) At:拟南芥;Os:水稻;PH:毛竹。下同。
Fig. 3 Phylogenetic tree(A),conserved motif(B)and conserved domains(C)in PheSLAC gene family At:Arabidopsis thaliana;Os:Oryza sativa;PH:Phyllostachys edulis. The same below.

图4 PheSLAC1蛋白关键氨基酸位点 橙色标记代表磷酸化位点,红色标记代表离子选择位点,绿色标记代表苯环。
Fig. 4 Key amino acids sites on PheSLAC1 proteins The orange label represents the phosphorylation site,the red label represents the ion selection site,and the green label represents the benzene ring.

图7 毛竹PheSLAC1过表达转基因株系的鉴定 拟南芥野生型Col和突变体slac1-3用作对照,肌动蛋白2用作内标。
Fig. 7 Characterization of PheSLAC overexpression transgenic lines Arabidopsis wild type Col and slac1-3 were used as the control,ACTIN 2 was used as an internal standard.
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
doi: 10.1093/mp/ssm022 pmid: 19825533 |
| [38] |
|
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
|
程明圣, 邹欢欢, 黄孝风, 陈慧晶, 邹娜, 杨清培. 2021. 干旱与盐胁迫对毛竹幼苗生长及生理的影响. 江西农业大学学报, 43 (4):842-852.
|
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
doi: 10.1111/nph.13435 pmid: 25932909 |
| [12] |
doi: 10.1111/nph.14685 pmid: 28722226 |
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
doi: 10.1016/j.cub.2015.01.067 pmid: 25802151 |
| [21] |
|
| [22] |
doi: 10.1093/pcp/pcf081 pmid: 12091719 |
| [23] |
|
| [24] |
|
| [25] |
|
| [39] |
|
|
应叶青, 郭璟, 魏建芬, 姜琴, 解楠楠. 2011. 干旱胁迫对毛竹幼苗生理特性的影响. 生态学杂志, 30 (2):262-266.
|
|
| [40] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
pmid: 16101906 |
| [31] |
pmid: 12232336 |
| [32] |
|
| [33] |
|
| [1] | 武祥琪, 孙鹂, 俞浙萍, 俞沁佩, 梁森苗, 郑锡良, 戚行江, 张淑文. 杨梅MrSPL4在响应干旱和低温胁迫中的作用研究[J]. 园艺学报, 2024, 51(5): 927-938. |
| [2] | 陈 鑫, 邬晓龙, 刘升锐, 胡贤春, 刘春艳, . 干旱胁迫下AMF对茶树光合特性及其基因表达的影响[J]. 园艺学报, 2024, 51(10): 2358-2370. |
| [3] | 田洁, 周倩怡, 铁原毓, 孙海宏, 黄思杰. 干旱胁迫下大蒜幼苗的代谢组学分析[J]. 园艺学报, 2024, 51(1): 133-144. |
| [4] | 王亦栖, 颜爽爽, 余炳伟, 甘雨薇, 邱正坤, 朱张生, 陈长明, 曹必好. 茄子青枯病抗性相关的E3泛素连接酶基因的筛选及鉴定[J]. 园艺学报, 2023, 50(10): 2271-2287. |
| [5] | 邱子文, 刘林敏, 林永盛, 林晓洁, 李永裕, 吴少华, 杨超. 千层金MbEGS基因的克隆与功能分析[J]. 园艺学报, 2022, 49(8): 1747-1760. |
| [6] | 彭轶, 李元慧, 杨瑞, 张子怡, 李亚楠, 韩云昊, 赵文超, 王绍辉. 茉莉酸合成基因LoxD参与调控番茄的抗旱性[J]. 园艺学报, 2022, 49(2): 319-331. |
| [7] | 张倩, 杨楠, 桑海煜, 赵荣, 宋晓惜, 陈龙清, 向林, 赵凯歌. 蜡梅花青素苷合成相关基因CpTT8的克隆和功能分析[J]. 园艺学报, 2021, 48(10): 1945-1955. |
| [8] | 张仲兴, 程丽, 王双成, 张德, 刘兵, 王延秀. 垂丝海棠MhMYB114-Like的克隆及其在苹果愈伤组织中的抗缺铁功能鉴定[J]. 园艺学报, 2021, 48(1): 127-136. |
| [9] | 黄尧瑶1,邓明华2,彭春秀2,文锦芬1,*. 百合花瓣抗氧化酶系统对干旱胁迫响应的研究[J]. 园艺学报, 2020, 47(4): 788-796. |
| [10] | 刘 偲 1,4,*,席 婉 1,2,*,袁金梅 1,2,朱琳琳 1,2,陈洪国,3 邹晶晶 3,郑日如 1,2,**,王彩云. 桂花‘莲籽丹桂’芳樟醇合酶基因 OfTPS5 的克隆及功能鉴定[J]. 园艺学报, 2020, 47(2): 310-320. |
| [11] | 宋 杨,刘红弟,王海波,张红军*,刘凤之*. 越橘花青苷合成相关基因VcTTG1的克隆与功能鉴定[J]. 园艺学报, 2019, 46(7): 1270-1278. |
| [12] | 杨官显,许海峰,张 静,王 楠,房鸿成,姜生辉,王意程,苏梦雨,陈学森*. 苹果花青苷调控基因MdMYB111的功能鉴定[J]. 园艺学报, 2019, 46(5): 832-840. |
| [13] | 王孝娣,冀晓昊,郑晓翠,王莹莹,宋 杨*,刘凤之*. 桃冷处理响应基因PdCIbHLH的克隆和功能鉴定[J]. 园艺学报, 2019, 46(3): 444-452. |
| [14] | 彭丽云,王 云,孙雪丽,王 晓,赵春丽,王丛巧,项蕾蕾,陈家兰,赖钟雄*,刘生财*. 苋菜中甜菜红素代谢相关基因AmMYB2的表达与功能分析[J]. 园艺学报, 2019, 46(3): 473-485. |
| [15] | 王春夏,尹 玥,王志平,严 瑞,付麟岚,孙红梅*. 多倍化兰州百合和细叶百合组培苗再生和耐非生物胁迫能力[J]. 园艺学报, 2019, 46(12): 2359-2368. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||
版权所有 © 2012 《园艺学报》编辑部 京ICP备10030308号-2 国际联网备案号 11010802023439
编辑部地址: 北京市海淀区中关村南大街12号中国农业科学院蔬菜花卉研究所 邮编: 100081
电话: 010-82109523 E-Mail: yuanyixuebao@126.com
技术支持:北京玛格泰克科技发展有限公司